GUÍA METODOLÓGICA DE ACÁMICA
Proyecto 03 Aplicaciones actuales
Resumen del proyecto
¡Aplica Procesamiento del Lenguaje Natural, Sistemas de Recomendación y Series de Tiempo para resolver problemas de relevancia contemporánea!
La realización y entrega del Proyecto es individual.
Entregables
Un Notebook de Jupyter con la resolución de la consigna. El Notebook debe poder ejecutarse sin errores. En el notebook debe estar el link al repositorio (por ejemplo, de GitHub) donde se pueda encontrar el proyecto entregado.
Referencias
Apóyate en las bitácoras, los notebooks trabajados y las presentaciones vistas en clase para resolver tu proyecto. También será de mucha utilidad la documentación de las librerías de Python. No dudes en consultar comunidades online como Stack Overflow y, por supuesto, buscar en la web (googlear).
Consigna
Elige una de las tres opciones de aplicación para elaborar tu proyecto. El objetivo es que apliques las herramientas aprendidas en el dominio que hayas seleccionado. Verás que para cualquiera de las opciones, el trabajo se organiza en tres partes:
- Parte A - Exploración de Datos: Todo proyecto de Ciencia de Datos empieza con un Análisis Exploratorio de Datos. Y todo Análisis Exploratorio de Datos debe responder preguntas.
- Parte B - Modelo de Machine Learning: En esta sección deberás aplicar las técnicas de Machine Learning aprendidas para crear un modelo predictivo a partir del dataset provisto.
- Parte C - Investigación: Las preguntas y cosas para probar nunca se agotan. El objetivo de esta sección es que sugieras cómo continuarías el proyecto, con el fin de mejorar el modelo o responder una pregunta que consideres interesante. En todos los notebooks dejamos algunas sugerencias, pero puedes proponer otras.
Checklist de evaluación
Sugerencias para desarrollar el proyecto:
La resolución del proyecto te puede enfrentar a desafíos que no trabajamos durante los encuentros. Es importante que desarrolles la capacidad de resolverlos. Para ello, consulta las bitácoras, las referencias, los notebooks vistos en clase, la documentación de la librería y, sobre todo, googlea.
- Los pasos deben estar correctamente justificados.
- Las preguntas que se respondan deben estar correctamente explicitadas.
- Imagina que este proyecto lo usarías para presentar en una entrevista de trabajo, o que lo debes presentar en tu trabajo. Presta mucha atención a la redacción, presentación de gráficos, etc.
Antes de subir tu proyecto a la plataforma Acámica para que sea evaluado, verifica que el Notebook se ejecute sin errores. Además, asegúrate de cumplir con las siguientes condiciones (son las que los/as evaluadores/as tendrán en consideración al momento de corregir tu trabajo):
Parte A - Exploración de Datos
- El Análisis Exploratorio de Datos debe servir para comprender el dataset y todo el flujo de trabajo que le siga.
- Debes responder al menos una pregunta original con este dataset. La pregunta debe estar correctamente explicitada.
Parte B - Modelo de Machine Learning
- Debes evaluar correctamente el modelo que realices. Esto implica un correcto manejo de datos de Train y Test, elegir una métrica apropiada y justificar su elección, y comparar los resultados contra un modelo benchmark.
- Puedes aplicar más de una de las técnicas vistas para crear tus modelos. Pero ten en cuenta que es preferible un modelo bien hecho (apropiada transformación de datos, optimización de hiperparámetros y análisis de sus resultados) que muchos modelos a medias. En caso de entrenar más de un modelo, debes comparar sus resultados y justificar cuál elegirías.
- Si el modelo lo permite, debes explorar qué información utiliza para predecir e interpretar ese resultado. ¿Coincide con lo que esperabas a partir de tu experiencia con el dataset?
Parte C - Investigación
- Debes explicar qué te gustaría probar, por qué y cómo lo harías. Si tienes referencias (por ejemplo, un artículo que hayas encontrado, capítulo de libro, etc.), debes mencionarlas.
- Debes comentar también qué resultados esperas encontrar. Por ejemplo, puedes implementar una prueba rápida y mostrar resultados preliminares, para ver si estás correctamente orientado. __ __
PROYECTO 3: ANÁLISIS TEMPORAL DE LOS CASOS DE DENGUE ENTRE 2010-2018 EN MEDELLÍN
Presentado por David Serna Gutiérrez.
Notebook disponible en el repositorio de Git-Hub: https://github.com/dsernag/Data_Science_Acamica
Ingeniero Forestal de la Universidad Nacional de Colombia Sede Medellín
Estudiante de Especialización en Sistemas de Información Geográfica
© Todos los derechos reservados ___
PARTE A - EXPLORACIÓN DE DATOS
La siguiente base de datos está disponbile en el enlace: http://medata.gov.co/dataset/dengue
Corresponde al registro de pacientes atendidos en las Instituciones Prestadoras de Servicios de Salud con diagnóstico probable o confirmado de Dengue y notificados al Sistema Nacional de Vigilancia en Salud Pública (SIVIGILA) desde el año 2008 al 2018. Para el estudio de series de tiempo en particular, fec_con es la variable fundamental para construir un data frame apropiado.
SECCIÓN A.I - DEPURACIÓN DE INFORMACIÓN
import numpy as np
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
import warnings
import timeit
warnings.filterwarnings('ignore')
url_dengue = "http://medata.gov.co/node/19391/download"
dengue_raw = pd.read_csv(url_dengue,encoding='utf-8',delimiter=";")
#Para corregir un poco el nombre de las columnas:
columnas = []
for i, elemnt in enumerate(dengue_raw.columns):
columnas.append(elemnt.split(".")[1])
dengue_raw.columns = columnas
dengue_raw.head()
| id | semana | edad_ | uni_med_ | sexo_ | nombre_barrio | comuna | tipo_ss_ | cod_ase_ | fec_con_ | ... | somnolenci | hipotensio | hepatomeg | hem_mucosa | hipotermia | aum_hemato | caida_plaq | acum_liquievento | evento | year_ | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | 24 | 49.0 | 1 | M | Belen | Belen | C | EAS022 | 19/06/2010 | ... | SD | SD | SD | SD | SD | SD | SD | SD | DENGUE | 2010 |
| 1 | 2 | 24 | 47.0 | 1 | M | Belen | Belen | C | CCF002 | 18/06/2010 | ... | SD | SD | SD | SD | SD | SD | SD | SD | DENGUE | 2010 |
| 2 | 3 | 24 | 46.0 | 1 | M | Sucre | Villa Hermosa | C | EPS010 | 21/06/2010 | ... | SD | SD | SD | SD | SD | SD | SD | SD | DENGUE | 2010 |
| 3 | 4 | 24 | 45.0 | 1 | M | SIN INFORMACION | SIN INFORMACION | C | EPS010 | 15/06/2010 | ... | SD | SD | SD | SD | SD | SD | SD | SD | DENGUE | 2010 |
| 4 | 5 | 24 | 46.0 | 1 | M | Perpetuo Socorro | La Candelaria | C | EPS010 | 20/06/2010 | ... | SD | SD | SD | SD | SD | SD | SD | SD | DENGUE | 2010 |
5 rows × 38 columns
#Partir la fecha de contagios en columnas independientes y que sean numéricas para ver su distribución
dengue_raw[['dia','mes','año']] = dengue_raw.fec_con_.str.split("/",expand=True)
#A números
#dengue_raw[['d_num','mes_num','año_num']] = (dengue_raw[['dia','mes','año']]).astype(int)
#Miramos la distribución:
dengue_raw.describe()
| id | semana | edad_ | cod_dpto_r | cod_mpio_r | year_ | |
|---|---|---|---|---|---|---|
| count | 51708.000000 | 51708.000000 | 51708.000000 | 51708.0 | 51708.0 | 51708.000000 |
| mean | 25854.500000 | 26.012222 | 30.899242 | 5.0 | 1.0 | 2013.340121 |
| std | 14926.958196 | 12.845455 | 18.749698 | 0.0 | 0.0 | 2.896628 |
| min | 1.000000 | 1.000000 | 0.000000 | 5.0 | 1.0 | 2008.000000 |
| 25% | 12927.750000 | 17.000000 | 16.000000 | 5.0 | 1.0 | 2010.000000 |
| 50% | 25854.500000 | 26.000000 | 28.000000 | 5.0 | 1.0 | 2014.000000 |
| 75% | 38781.250000 | 35.000000 | 44.000000 | 5.0 | 1.0 | 2016.000000 |
| max | 51708.000000 | 53.000000 | 174.000000 | 5.0 | 1.0 | 2018.000000 |
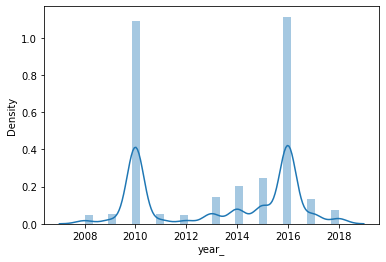
Al comparar el año extraido de la fecha de contagio con year, se observa que hay mucha dispariedad entre los valores:
print(f"Este es el año que trae el data set como 'year': \n\n: {dengue_raw.year_.value_counts().sort_index(ascending=True)}")
print("\n")
print(f"Este es el año que extraje de 'fec_con' es decir fecha de contagio: \n\n: {dengue_raw.año.value_counts().sort_index(ascending=True)}")
Este es el año que trae el data set como 'year':
: 2008 722
2009 842
2010 17641
2011 829
2012 726
2013 2294
2014 3311
2015 3998
2016 18003
2017 2154
2018 1188
Name: year_, dtype: int64
Este es el año que extraje de 'fec_con' es decir fecha de contagio:
: 1900 9
2004 1
2007 10
2008 704
2009 821
2010 17654
2011 832
2012 732
2013 2301
2014 3165
2015 3909
2016 18166
2017 2207
2018 1188
2019 9
Name: año, dtype: int64
La variable year viene por defecto en el dataset y representa el año de la observación, mientras año, extraido de fec_con (fecha de contagio), es la observación reportada ante el SIVIGILA. No obstante year presenta mejor distribución
sns.distplot(dengue_raw.año)
plt.show()

sns.distplot(dengue_raw.year_)
plt.show()
¿ Cuál escoger entonces? Al menos los días y los meses encajan (aparentemente, pues puede haber meses que tienen más meses de los que debería). Creo conveniente continuar con year. Esa será la variable a concatenar con mes y dia para crear una nueva variable date de tipo date:
#Pasar el año a string
dengue_raw['y_string'] = (dengue_raw['year_']).astype(str)
#Agregarlos en un objeto
dates=dengue_raw[['y_string', 'mes', 'dia']].agg('-'.join, axis=1)
#Pegarlos como fecha en `date`
dengue_raw['date']= pd.to_datetime(dates)
dengue_raw.head()
| id | semana | edad_ | uni_med_ | sexo_ | nombre_barrio | comuna | tipo_ss_ | cod_ase_ | fec_con_ | ... | aum_hemato | caida_plaq | acum_liquievento | evento | year_ | dia | mes | año | y_string | date | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | 24 | 49.0 | 1 | M | Belen | Belen | C | EAS022 | 19/06/2010 | ... | SD | SD | SD | DENGUE | 2010 | 19 | 06 | 2010 | 2010 | 2010-06-19 |
| 1 | 2 | 24 | 47.0 | 1 | M | Belen | Belen | C | CCF002 | 18/06/2010 | ... | SD | SD | SD | DENGUE | 2010 | 18 | 06 | 2010 | 2010 | 2010-06-18 |
| 2 | 3 | 24 | 46.0 | 1 | M | Sucre | Villa Hermosa | C | EPS010 | 21/06/2010 | ... | SD | SD | SD | DENGUE | 2010 | 21 | 06 | 2010 | 2010 | 2010-06-21 |
| 3 | 4 | 24 | 45.0 | 1 | M | SIN INFORMACION | SIN INFORMACION | C | EPS010 | 15/06/2010 | ... | SD | SD | SD | DENGUE | 2010 | 15 | 06 | 2010 | 2010 | 2010-06-15 |
| 4 | 5 | 24 | 46.0 | 1 | M | Perpetuo Socorro | La Candelaria | C | EPS010 | 20/06/2010 | ... | SD | SD | SD | DENGUE | 2010 | 20 | 06 | 2010 | 2010 | 2010-06-20 |
5 rows × 43 columns
Dado que en un futuro me interesaría ver el comportamiento del fenómeno en el espacio, desecharé aquellas observaciones que no tengan barrio o comuna.
#Primero pasaré `nombre_barrio` y `comuna` a mayúscula sostenida y remplazaré cualquier caracter especial por su homónimo:
caracteres_especiales = {'Á':'A','É':'E','Í':'I','Ó':'O','Ú':'U','Ñ':'N'}
for i,j in caracteres_especiales.items():
dengue_raw['comuna'] = dengue_raw['comuna'].str.upper().str.replace(i,j)
dengue_raw['nombre_barrio'] = dengue_raw['nombre_barrio'].str.upper().str.replace(i,j)
#Acá vemos todas las comunas reportados
for i in dengue_raw.comuna.sort_values().unique():
print(i)
ALTAVISTA
ARANJUEZ
BELEN
BUENOS AIRES
CASTILLA
CORREGIMIENTO DE SAN CRISTOBAL
CORREGIMIENTO DE SANTA ELENA
DOCE DE OCTUBRE
EL POBLADO
GUAYABAL
LA AMERICA
LA CANDELARIA
LAURELES
MANRIQUE
POPULAR
ROBLEDO
SAN ANTONIO DE PRADO
SAN JAVIER
SAN SEBASTIAN DE PALMITAS
SANTA CRUZ
SIN INFORMACION
VILLA HERMOSA
##Acá vemos todos los barrios reportados
#for i in dengue_raw.nombre_barrio.sort_values().unique():
# print(i)
dengue_raw.nombre_barrio.value_counts()
SIN INFORMACION 3208
SAN ANTONIO DE PRADO 1175
LAS GRANJAS 637
VILLATINA 630
SAN BERNARDO 575
...
URQUITA 1
SUBURBANO POTRERA MISERENGA 1
SUBURBANO EL PLACER 1
SUBURBANO PALMITAS 1
SUBURBANO URQUITA 1
Name: nombre_barrio, Length: 326, dtype: int64
dengue_raw.comuna.value_counts()
VILLA HERMOSA 4765
BELEN 4242
MANRIQUE 4149
BUENOS AIRES 3719
ARANJUEZ 3659
SAN JAVIER 3358
SIN INFORMACION 3190
ROBLEDO 2834
POPULAR 2642
DOCE DE OCTUBRE 2640
LA CANDELARIA 2595
SANTA CRUZ 2319
CASTILLA 1938
LA AMERICA 1883
LAURELES 1791
GUAYABAL 1577
EL POBLADO 1573
SAN ANTONIO DE PRADO 1222
ALTAVISTA 828
CORREGIMIENTO DE SAN CRISTOBAL 568
CORREGIMIENTO DE SANTA ELENA 193
SAN SEBASTIAN DE PALMITAS 23
Name: comuna, dtype: int64
Existen valores SIN INFORMACION en nombre_barrio y comuna. Serán eliminados:
dengue_raw2 = dengue_raw.loc[(dengue_raw['nombre_barrio'] != 'SIN INFORMACION') & (dengue_raw['comuna'] != 'SIN INFORMACION')]
print(f"Se eliminaron:{dengue_raw.shape[0]-dengue_raw2.shape[0]} observaciones")
Se eliminaron:3208 observaciones
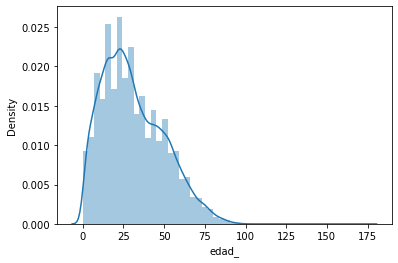
#Observaremos la edad y el sexo:
sns.distplot(dengue_raw2.edad_)
plt.show()
dengue_raw.edad_.value_counts().sort_index(ascending=False)
174.0 1
122.0 1
112.0 1
99.0 1
98.0 2
...
4.0 625
3.0 620
2.0 518
1.0 531
0.0 5
Name: edad_, Length: 103, dtype: int64
dengue_raw2.sexo_.value_counts().sort_index(ascending=False)
M 23613
F 24887
Name: sexo_, dtype: int64
Existen valores atípicos en la edad. Se desechará cualquier valor que supere 100 años. Y del sexo se eliminará la observación que tiene SD
dengue_raw3 = dengue_raw2.loc[dengue_raw['edad_'] < 100]
dengue_raw3.shape
(48497, 43)
print(f"Se eliminaron:{dengue_raw2.shape[0]-dengue_raw3.shape[0]} observaciones")
Se eliminaron:3 observaciones
Con los atributos más importantes corregidos, voy a escoger las variables para el estudio. En este caso, no nos interesa niveles de intensidad del virus, o síntomas. Cada observación es un caso de dengue con una estampa temporal date. Escogeré entonces:
dengue_tidy = dengue_raw3.iloc[:,[42,5,6,2,4]]
dengue_tidy.head()
| date | nombre_barrio | comuna | edad_ | sexo_ | |
|---|---|---|---|---|---|
| 0 | 2010-06-19 | BELEN | BELEN | 49.0 | M |
| 1 | 2010-06-18 | BELEN | BELEN | 47.0 | M |
| 2 | 2010-06-21 | SUCRE | VILLA HERMOSA | 46.0 | M |
| 4 | 2010-06-20 | PERPETUO SOCORRO | LA CANDELARIA | 46.0 | M |
| 5 | 2010-06-17 | TERMINAL DE TRANSPORTE | CASTILLA | 45.0 | M |
SECCIÓN A.II - INTERPRETACIÓN DE LOS DATOS
#Realizaré un análisis a nivel de comuna para obtener algunas conclusiones del fenómeno.
#Debo definiar una variable que contabilice la cantidad de casos. Esto para poder sumar los casos por comuna o barrio. Cada observación es un caso
#por lo que se asigna una columna contaste =1, así:
dengue_tidy['casos'] = 1
#Para tener una vista preliminar de cómo se comportaron los contagios entre 2010-2018
#Se hará una gráfica para cada uno a nivel:
#A nivel diario:
plt.figure(figsize=(24,10))
dengue_tidy.resample('D', on ='date').size().plot()
plt.ylabel('Casos diarios')
plt.title('Casos de dengue 2010-2018 resolución diaria')
#A nivel semanal:
plt.figure(figsize=(24,10))
dengue_tidy.resample('W', on ='date').size().plot()
plt.ylabel('Casos semanales')
plt.title('Casos de dengue 2010-2018 resolución semanal')
#A nivel mensual:
plt.figure(figsize=(24,10))
dengue_tidy.resample('M', on ='date').size().plot()
plt.ylabel('Casos mensuales')
plt.title('Casos de dengue 2010-2018 resolución mensual')
#A nivel anual:
plt.figure(figsize=(24,10))
dengue_tidy.resample('A', on ='date').size().plot()
plt.ylabel('Casos anuales')
plt.title('Casos de dengue 2010-2018 resolución anual')
plt.show()
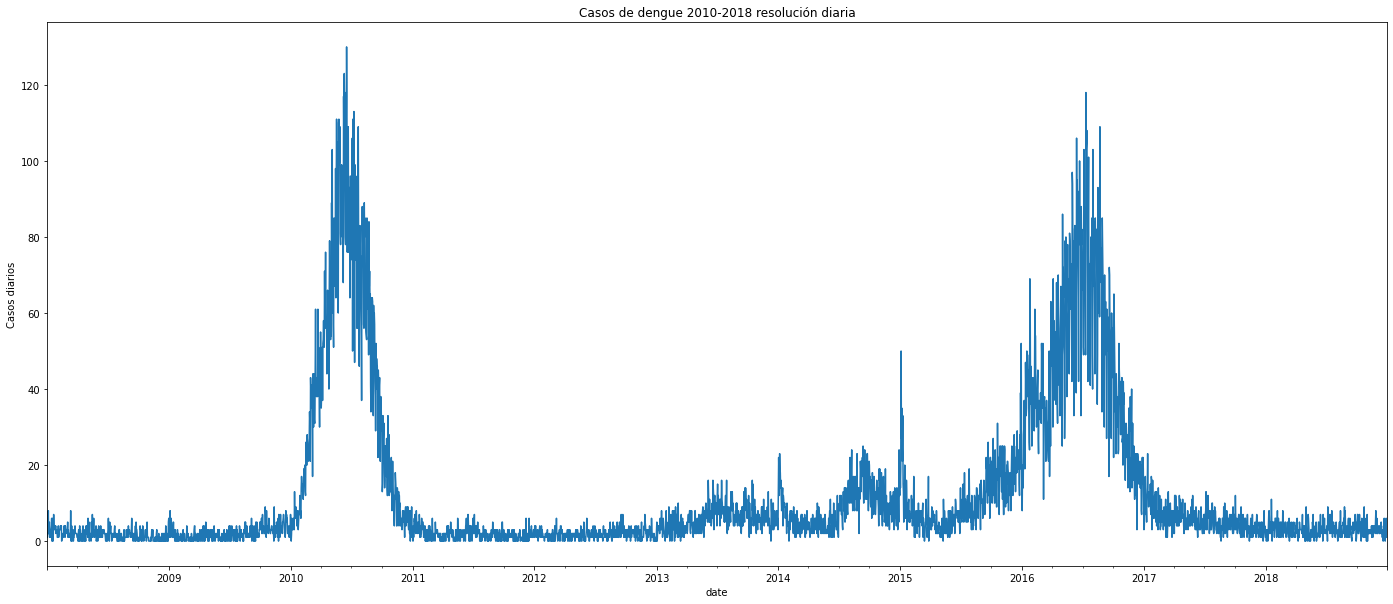
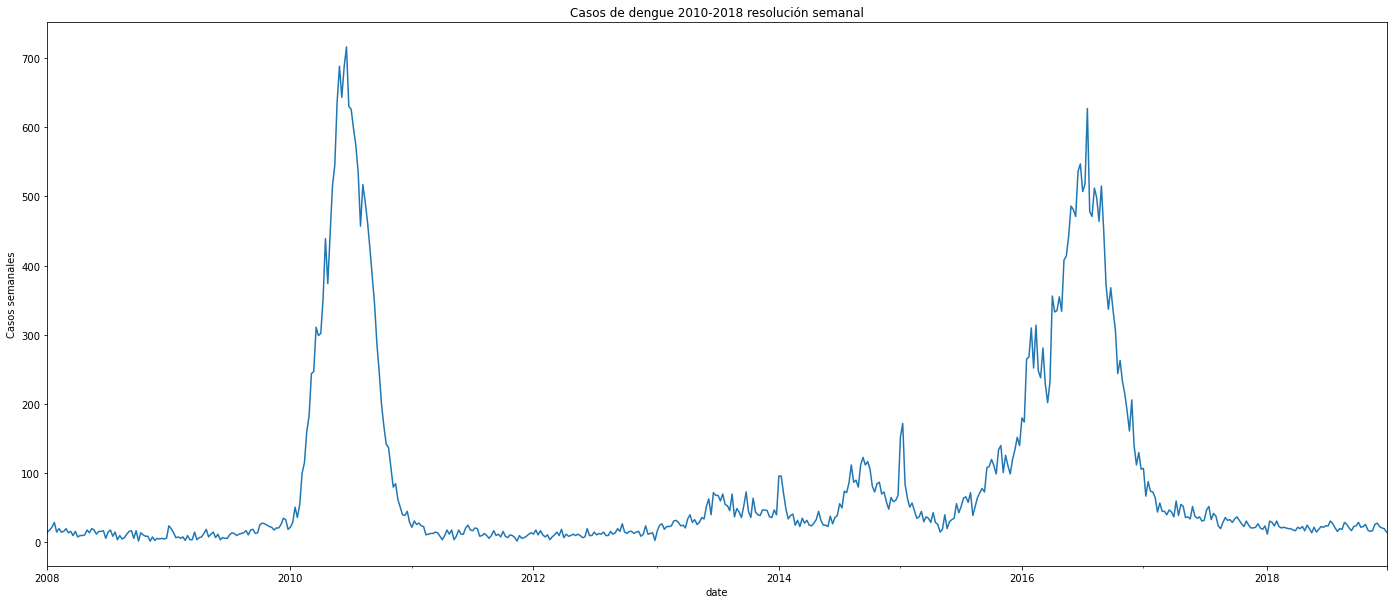
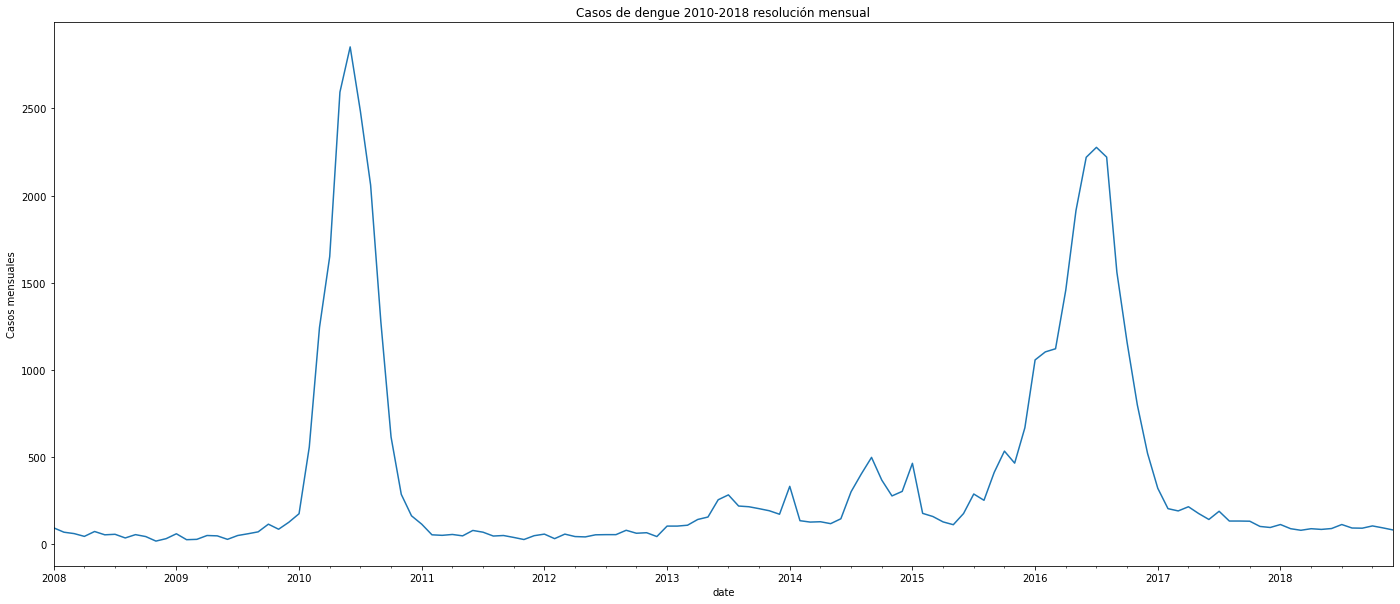
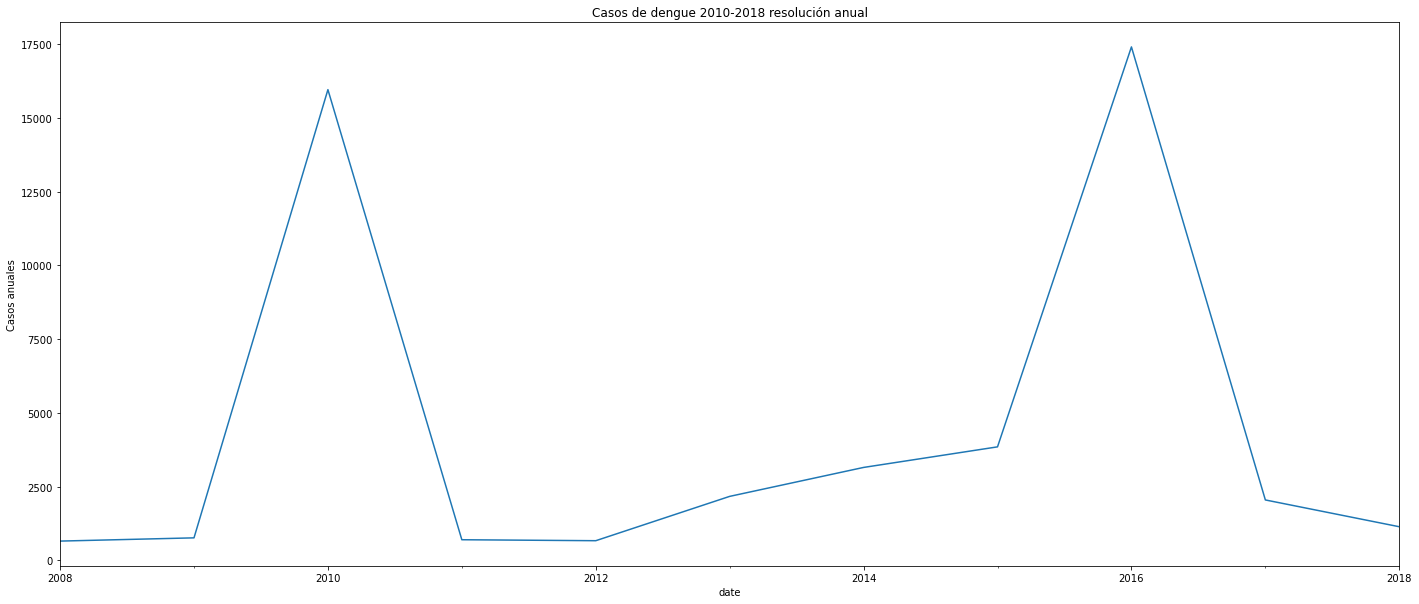
Al interpretar las figuras se observa que en 2010 hubo un pico de algo más de 15.000 casos en toda la ciudad. Desde 2012 hasta finales de 2015 se observa un incremento constante en los casos cada año, para en 2016 presentar un pico mucho más alto con casi 17.500 casos. En las secciones de más adelante analizaré con mayor detalle algún tipo de autocorrelación, e intentar predecir a futuro con base en el pasado.
#Al realizar este resample puedo encontrar la cantidad de casos por año:
print(dengue_tidy.resample('AS', on ='date').casos.sum().reset_index())
print("\nLos años con mayores contagios fueron 2010 y 2016, acá hay algún patrón estacional que debe ser investigado más adelante")
date casos
0 2008-01-01 650
1 2009-01-01 760
2 2010-01-01 15966
3 2011-01-01 696
4 2012-01-01 663
5 2013-01-01 2167
6 2014-01-01 3149
7 2015-01-01 3847
8 2016-01-01 17416
9 2017-01-01 2046
10 2018-01-01 1137
Los años con mayores contagios fueron 2010 y 2016, acá hay algún patrón estacional que debe ser investigado más adelante
A continuación crearé un dataset donde agruparé los casos por comuna por mes:
#Creamos el resampleo mensual de dengue por barrio:
dengue_comuna = dengue_tidy.groupby('comuna').resample('MS', on ='date').sum().reset_index()
dengue_comuna = dengue_comuna.set_index('date')
dengue_comuna = dengue_comuna.drop(['edad_'],axis=1)
#Volver a poner el índice de date:
dengue_comuna.head()
| comuna | casos | |
|---|---|---|
| date | ||
| 2008-05-01 | ALTAVISTA | 1 |
| 2008-06-01 | ALTAVISTA | 0 |
| 2008-07-01 | ALTAVISTA | 0 |
| 2008-08-01 | ALTAVISTA | 0 |
| 2008-09-01 | ALTAVISTA | 0 |
#En este gráfico podemos ver el comportamiento del fenómono por cada comuna
plt.figure(figsize=(24,10))
sns.lineplot(data=dengue_comuna, x='date',y='casos', hue='comuna',palette='gist_ncar')
plt.ylabel('Casos acumulados por mes')
plt.xlabel('Años')
plt.show()
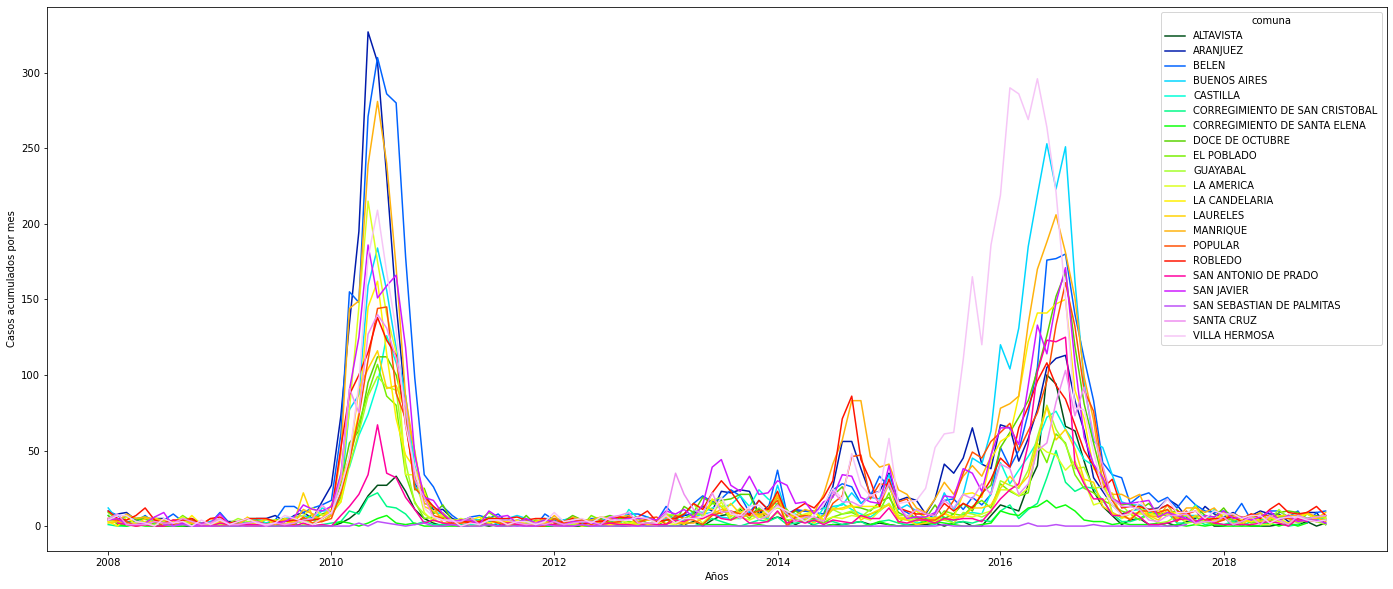
#Con el fin de obtener una mejor interpretación de cada comuna se graficará por cada comuna el comportamiento mensual de los contagios:
for comu in dengue_comuna.comuna.unique():
plt.figure(figsize=(24,10))
sns.lineplot(data=dengue_comuna.loc[dengue_comuna.comuna==comu], x='date',y='casos')
plt.ylabel('Casos acumulados por mes')
plt.xlabel('Años')
plt.title(comu,fontsize=25)
plt.show()
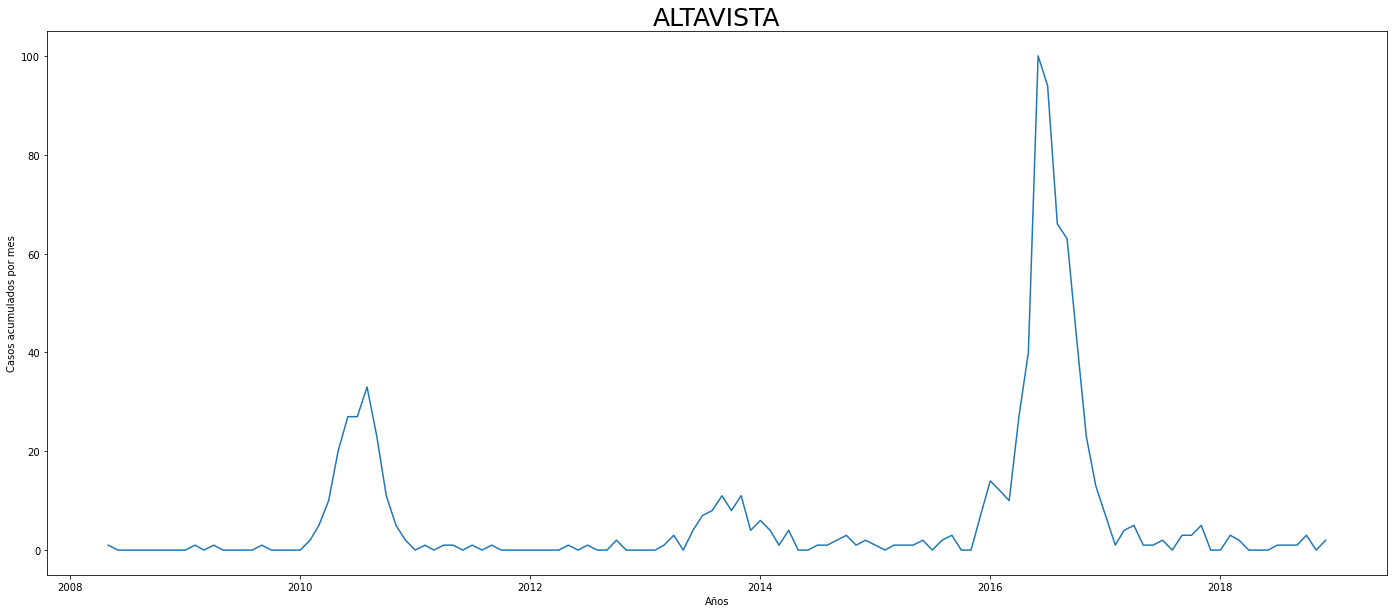
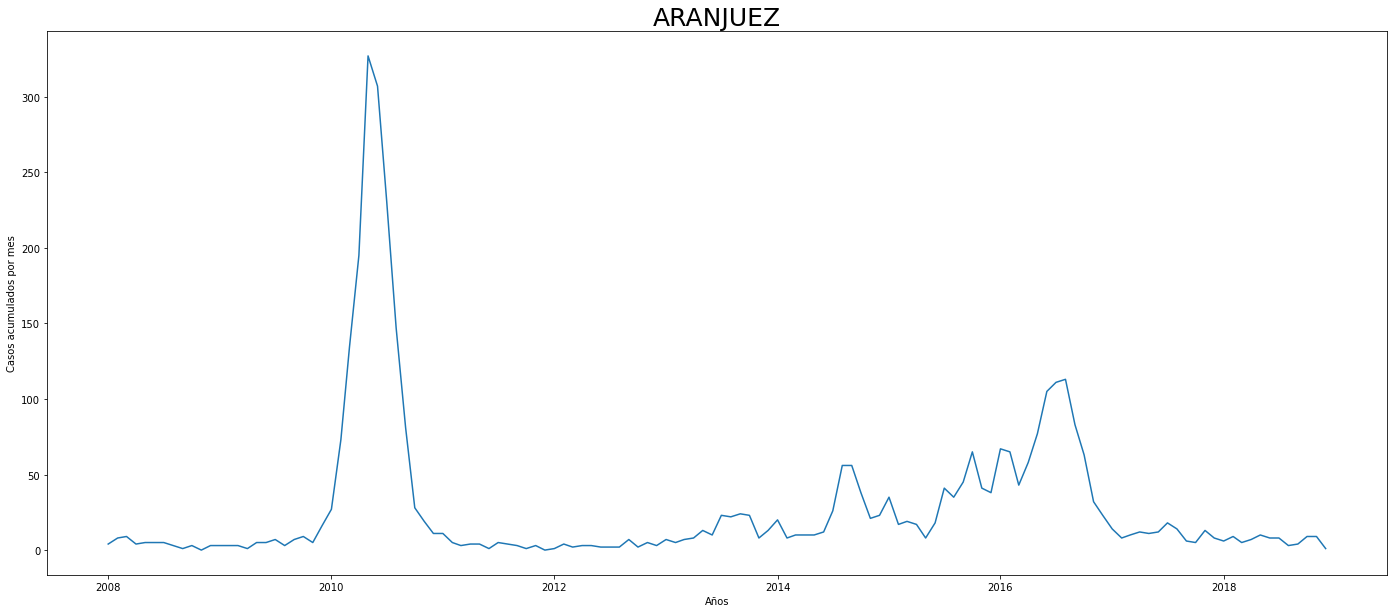
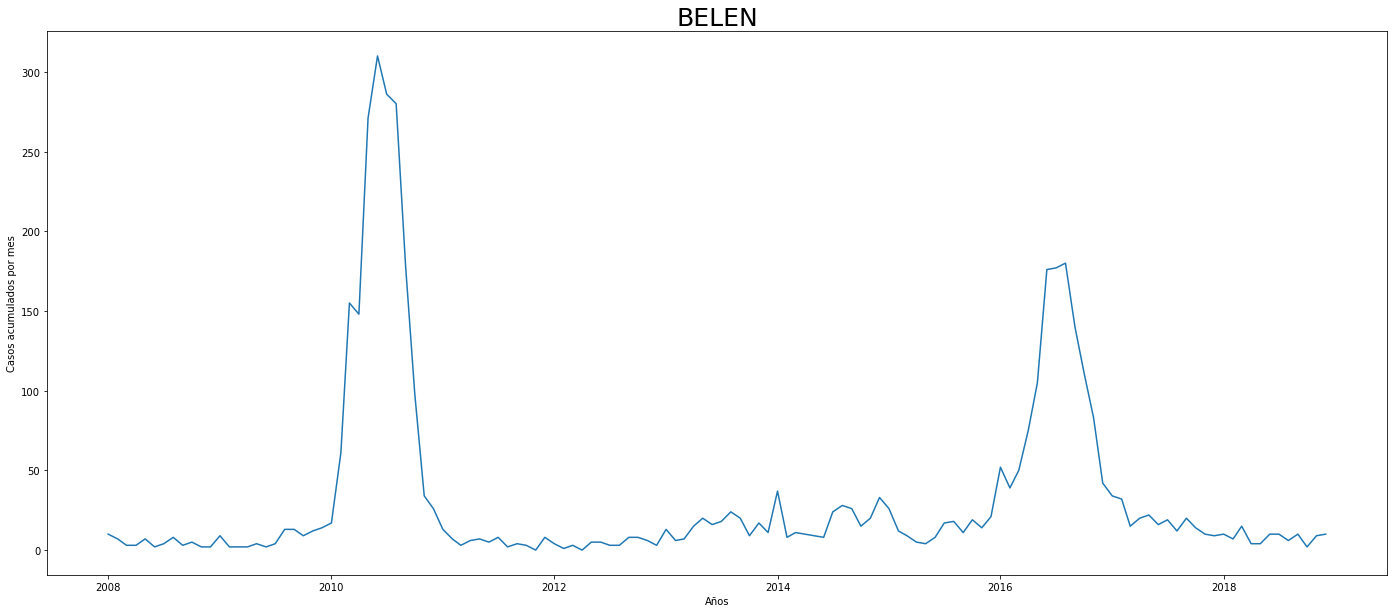
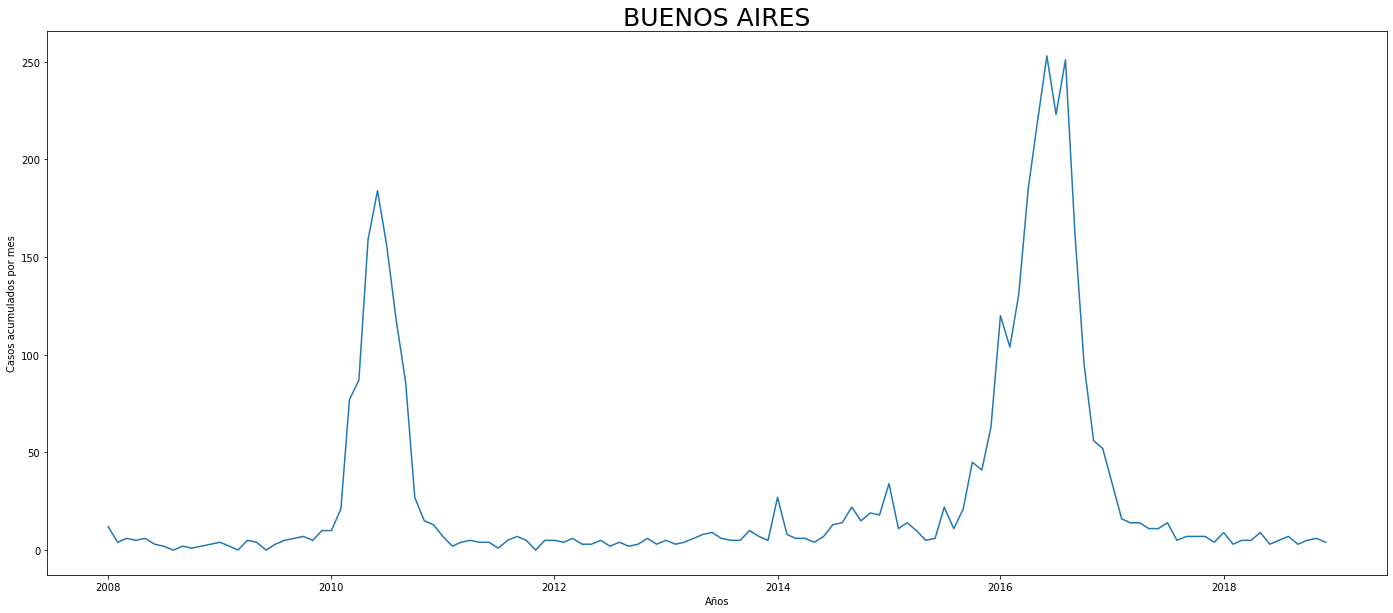
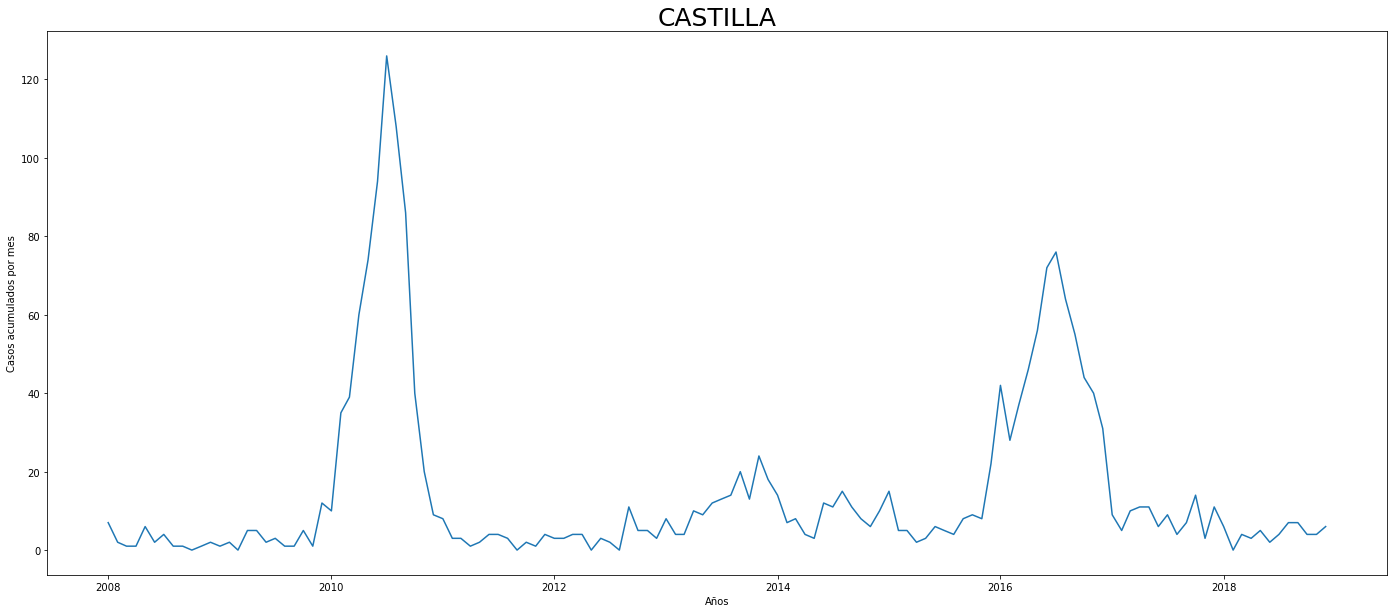
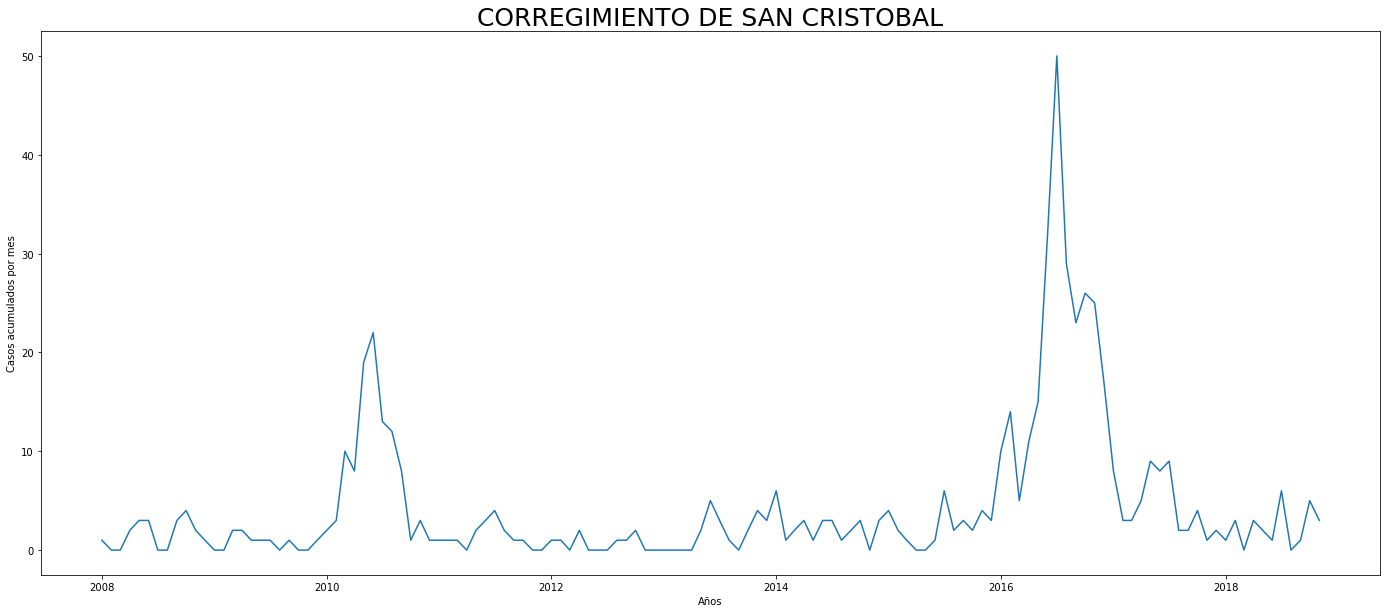
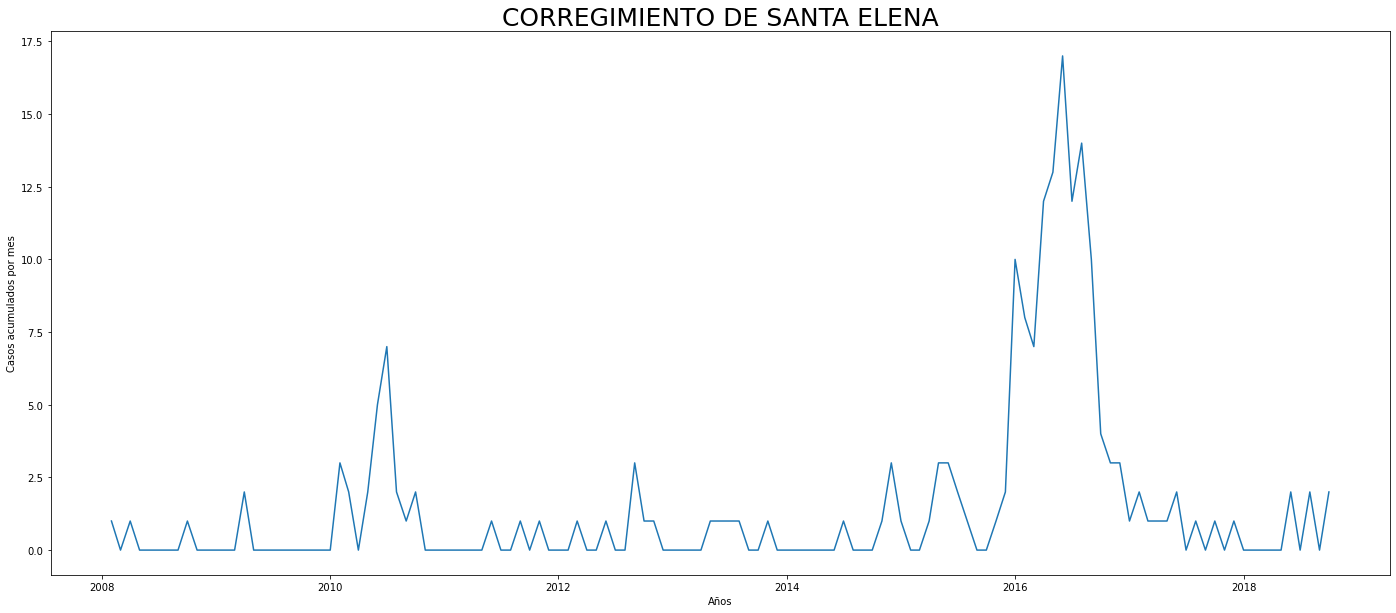
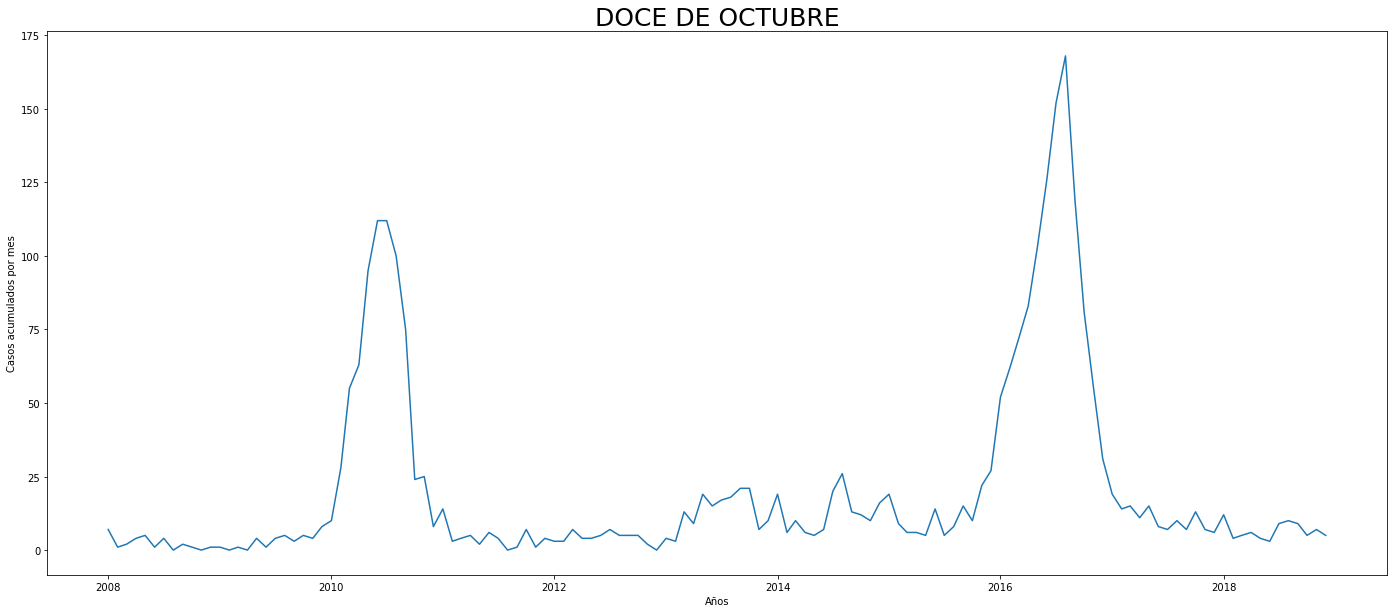
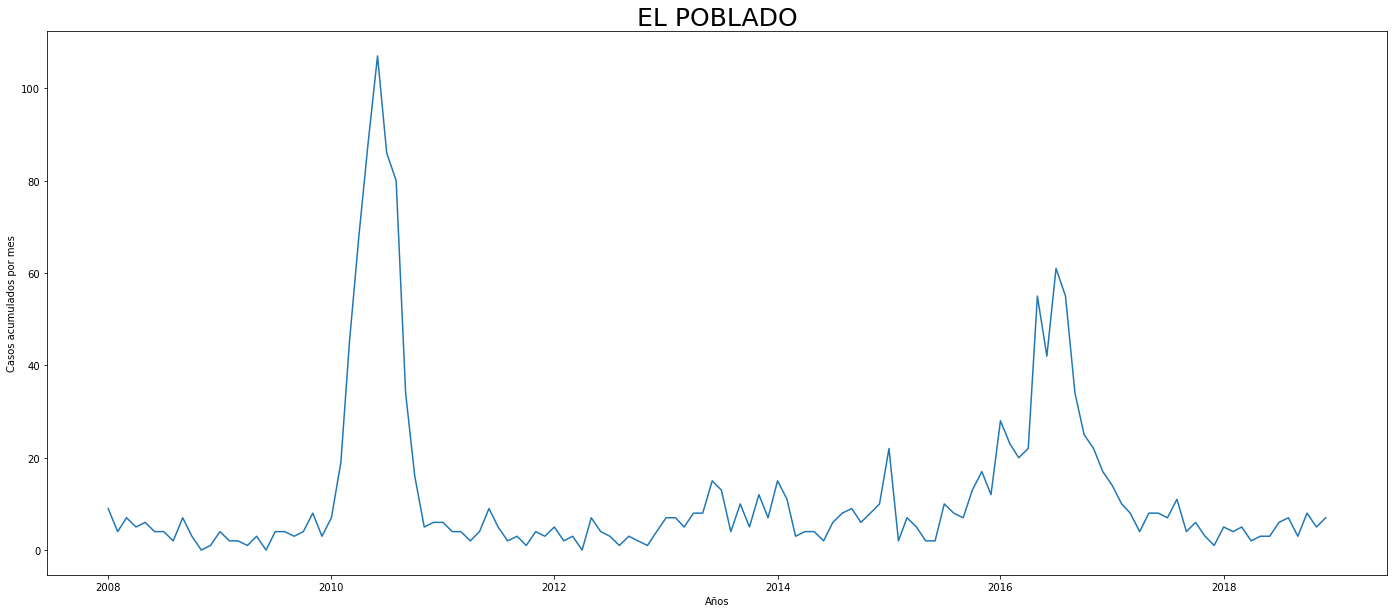
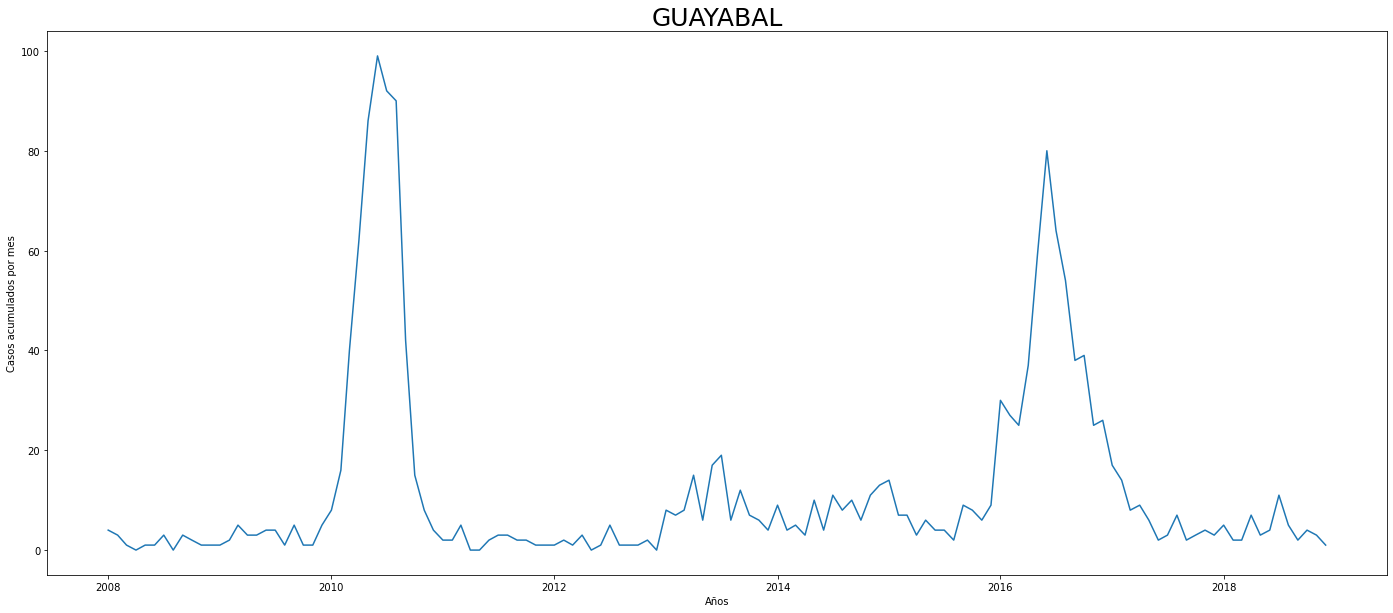
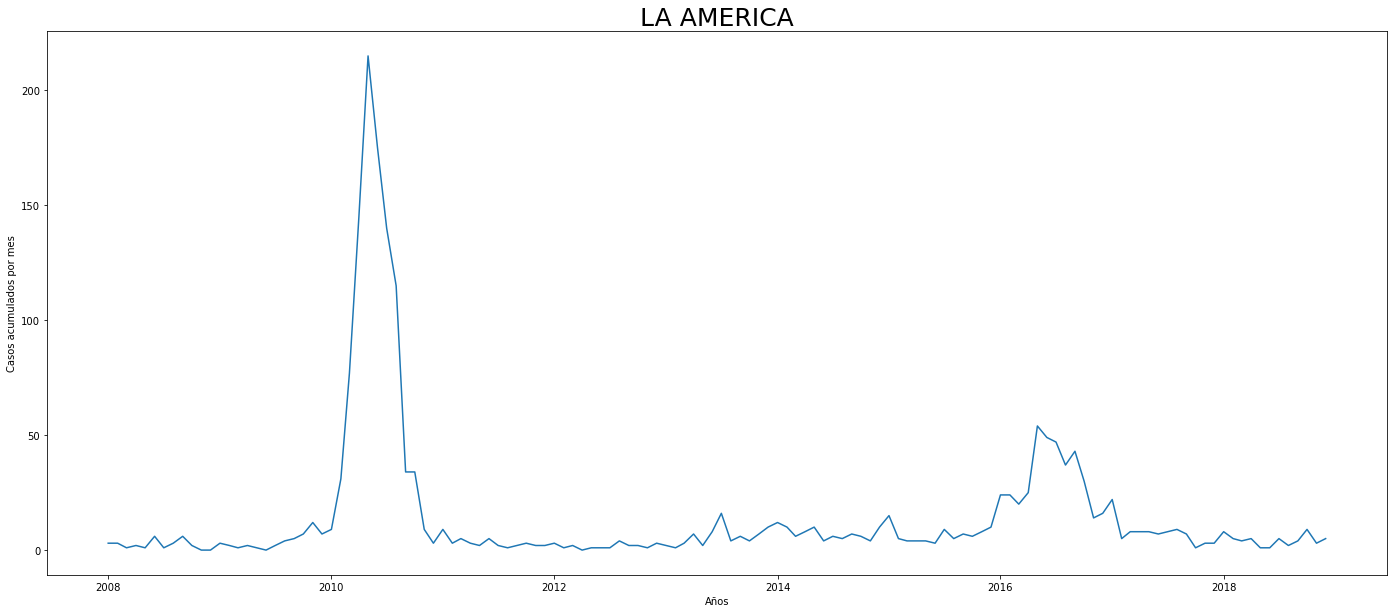
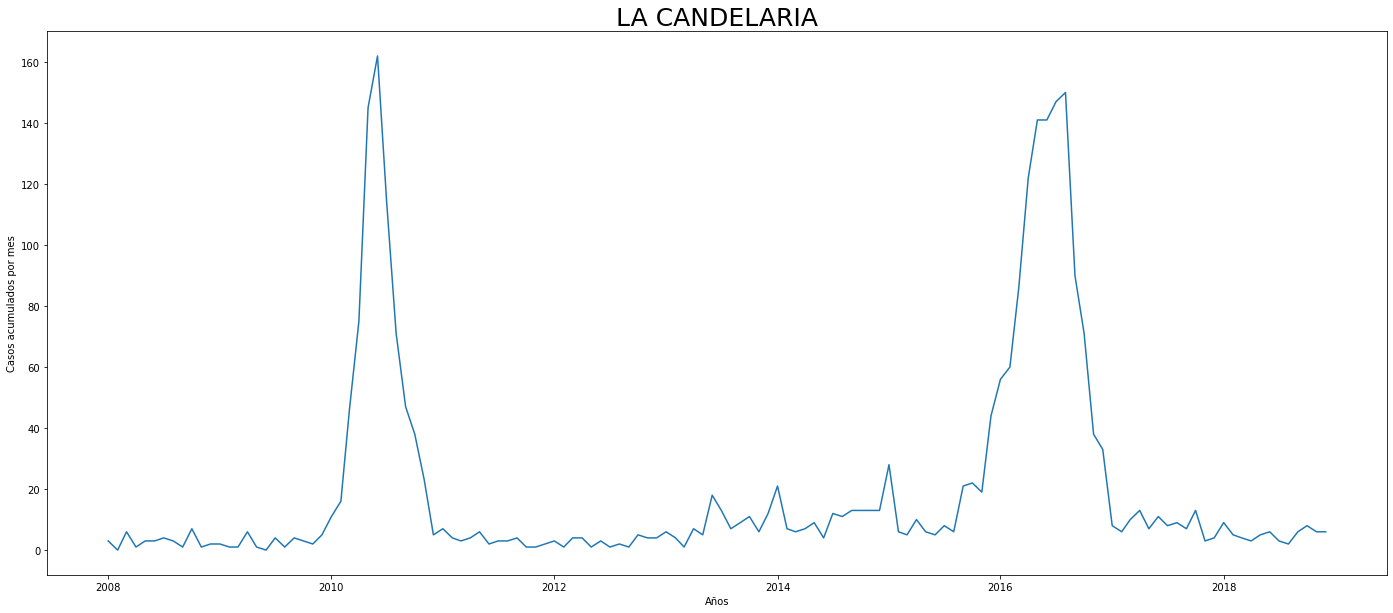
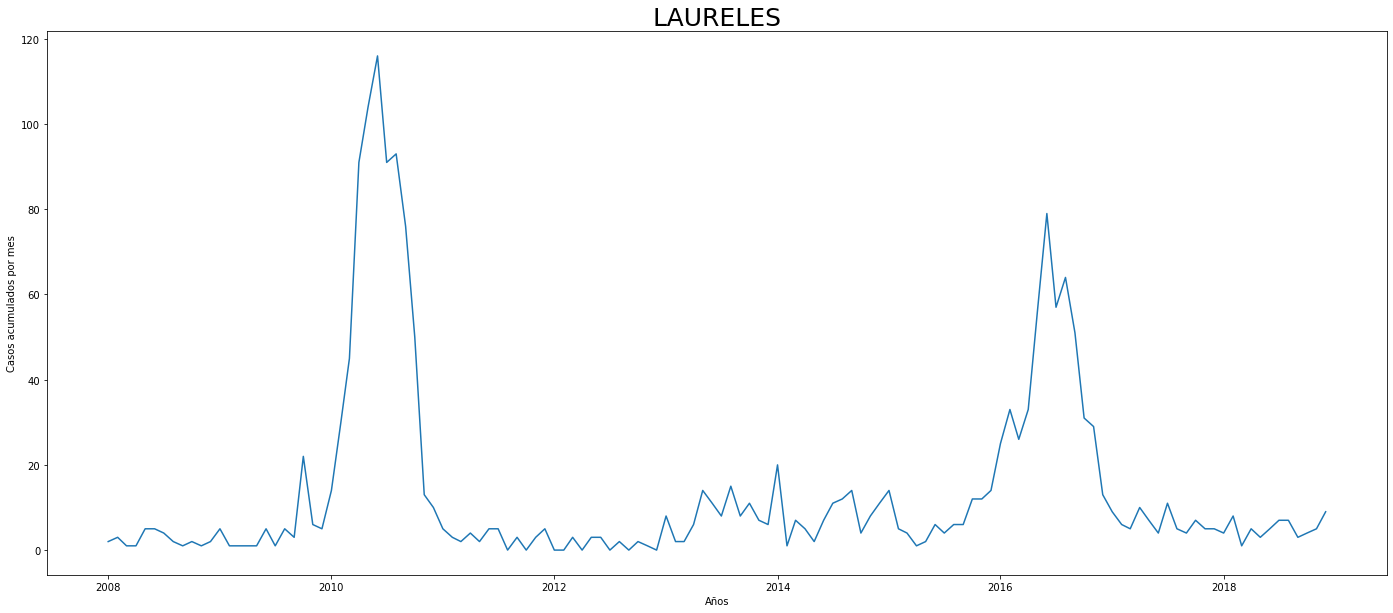
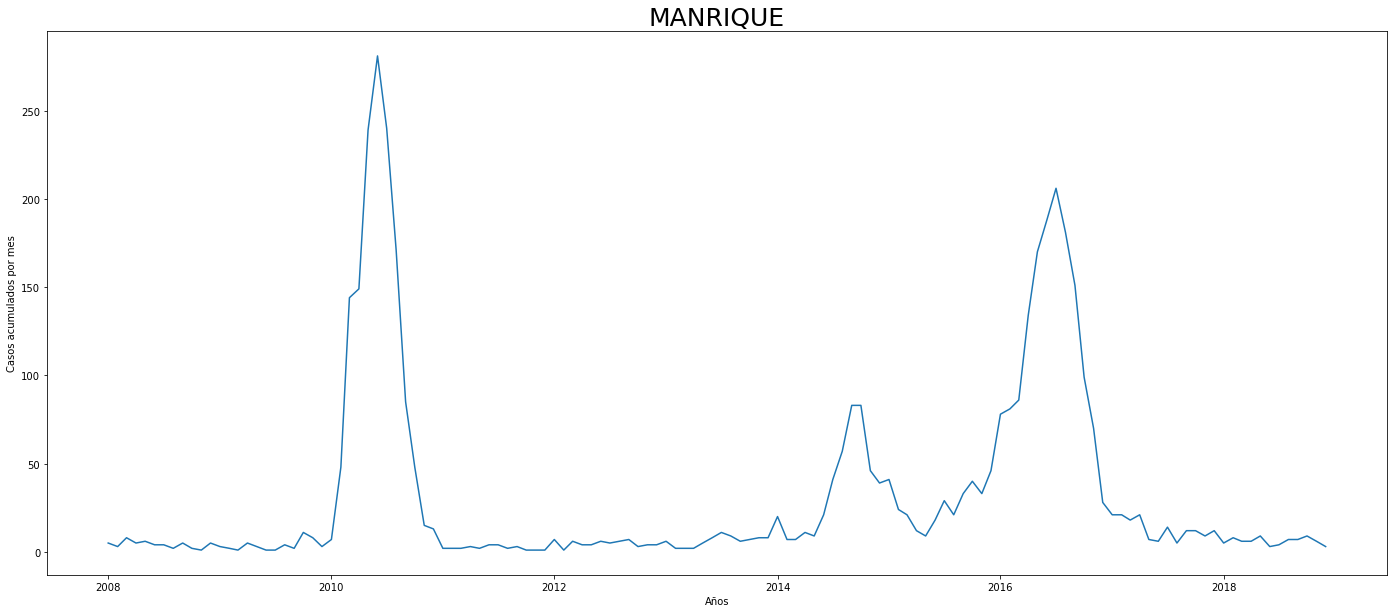
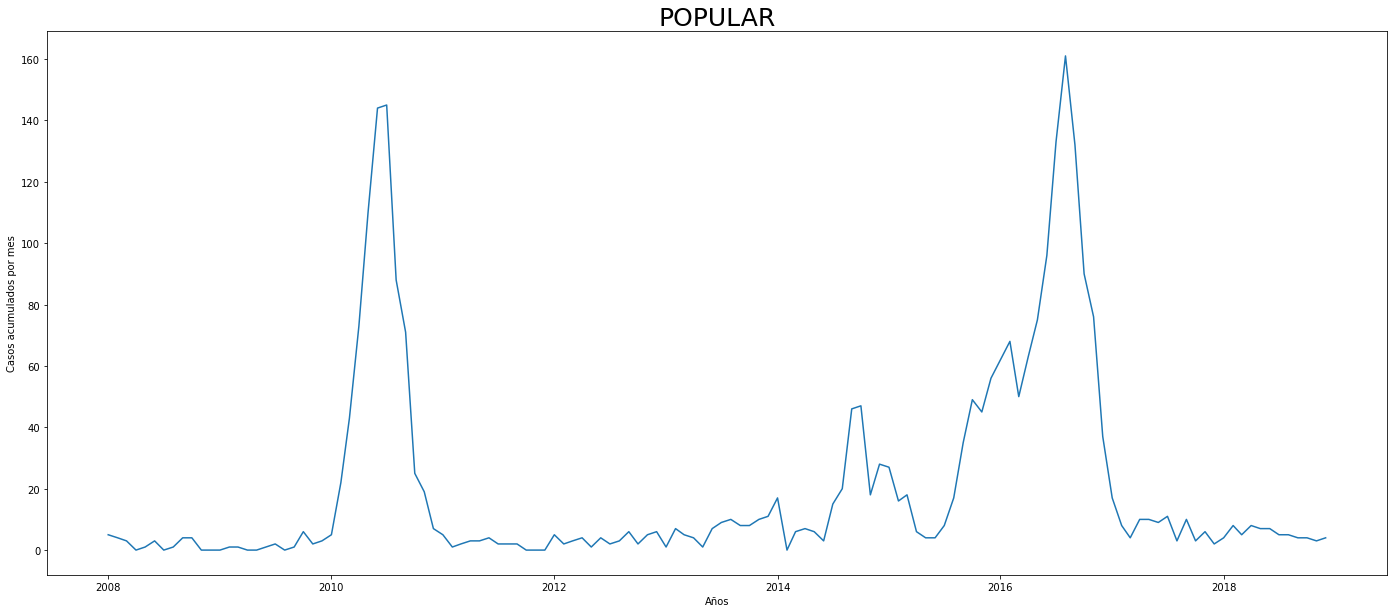
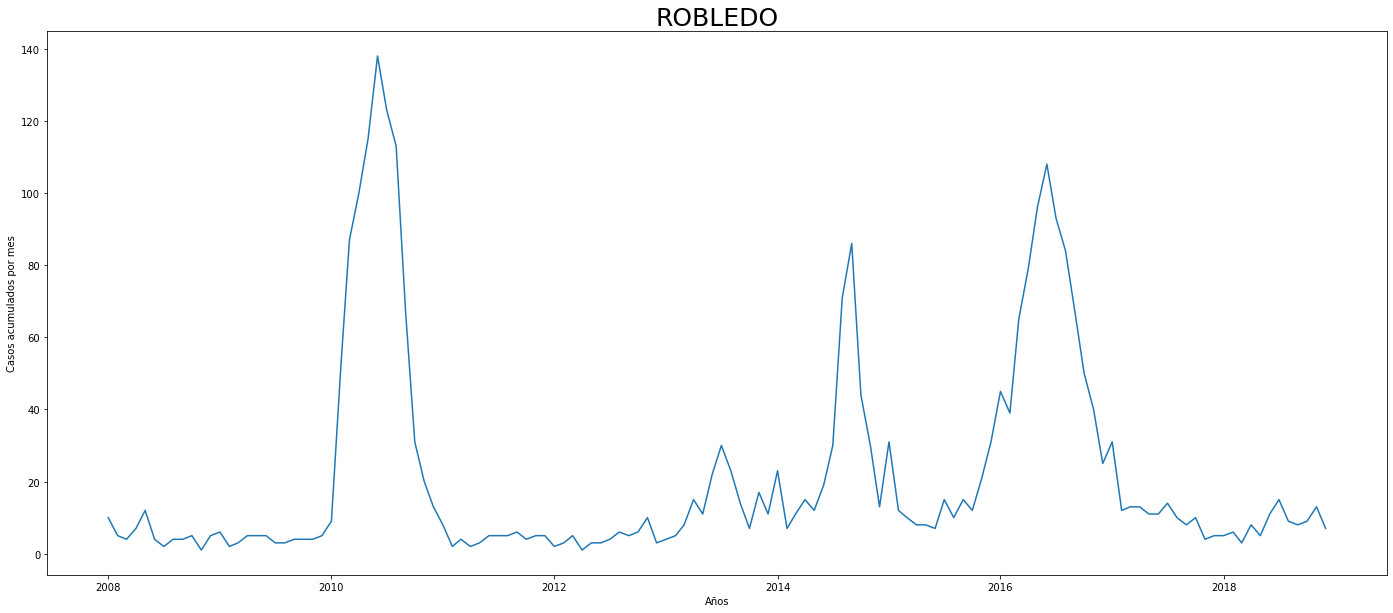
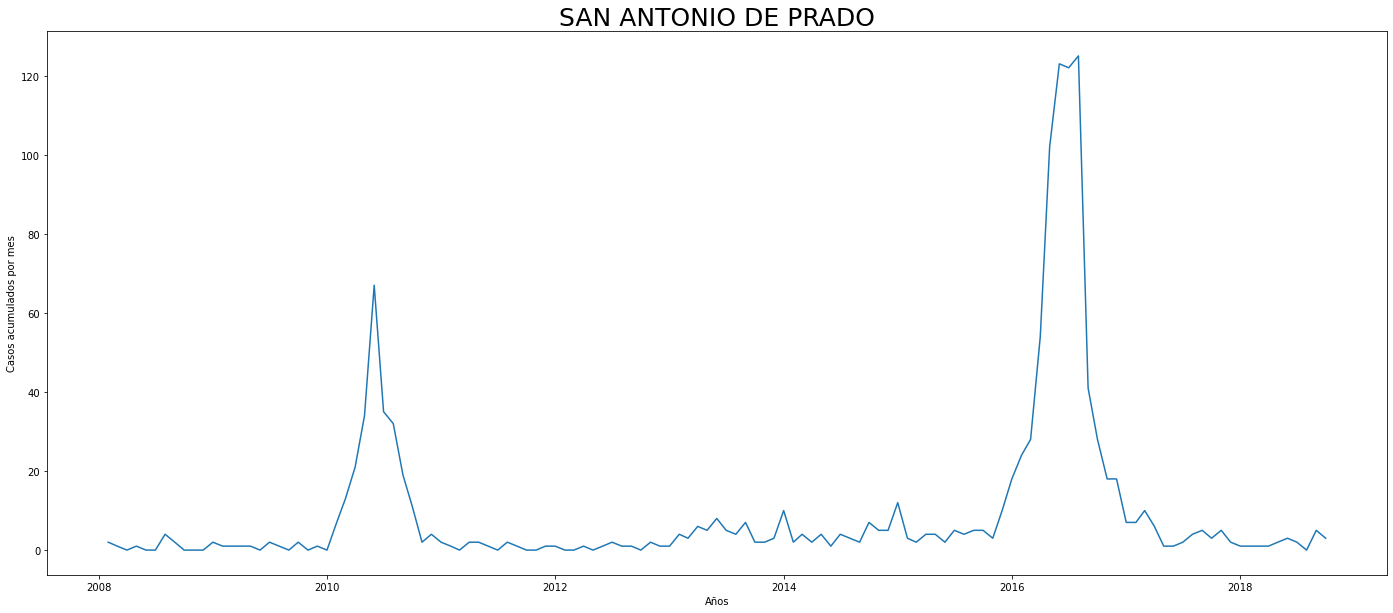
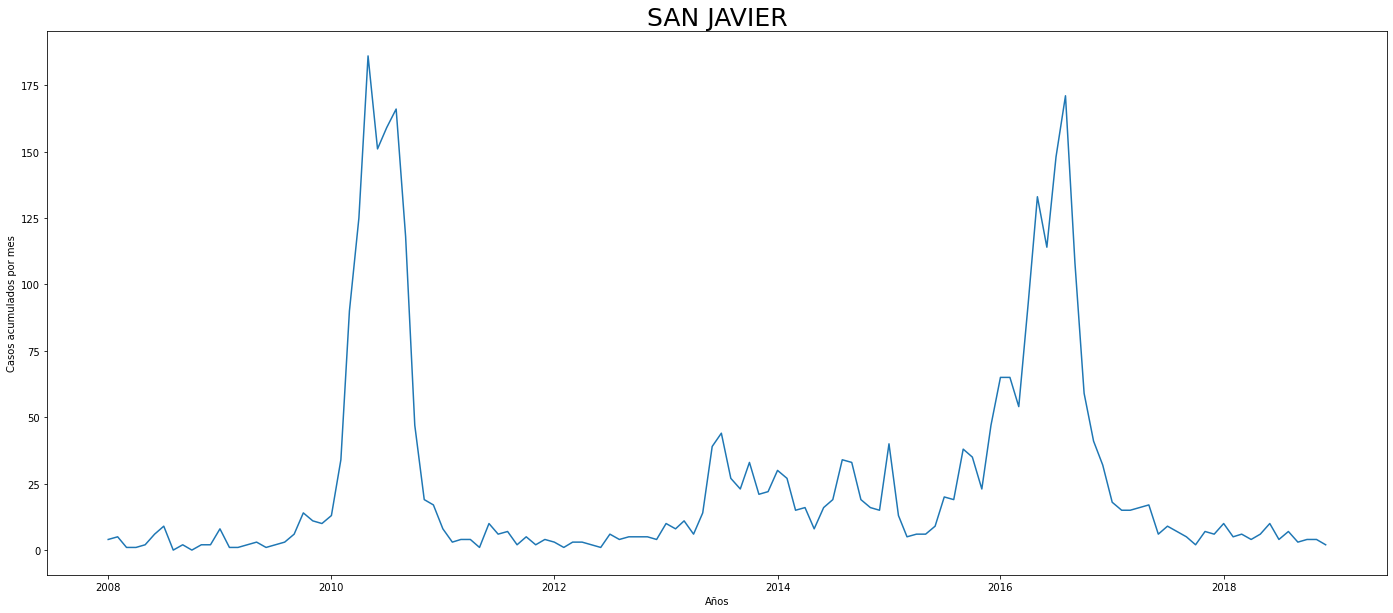
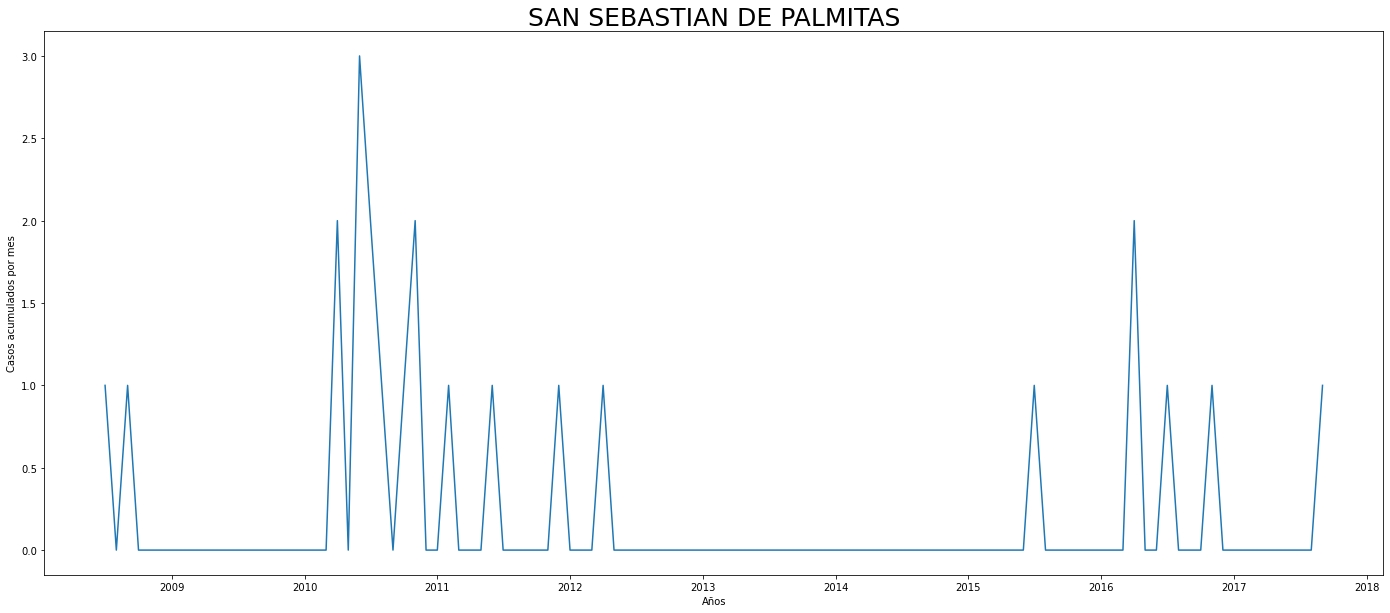
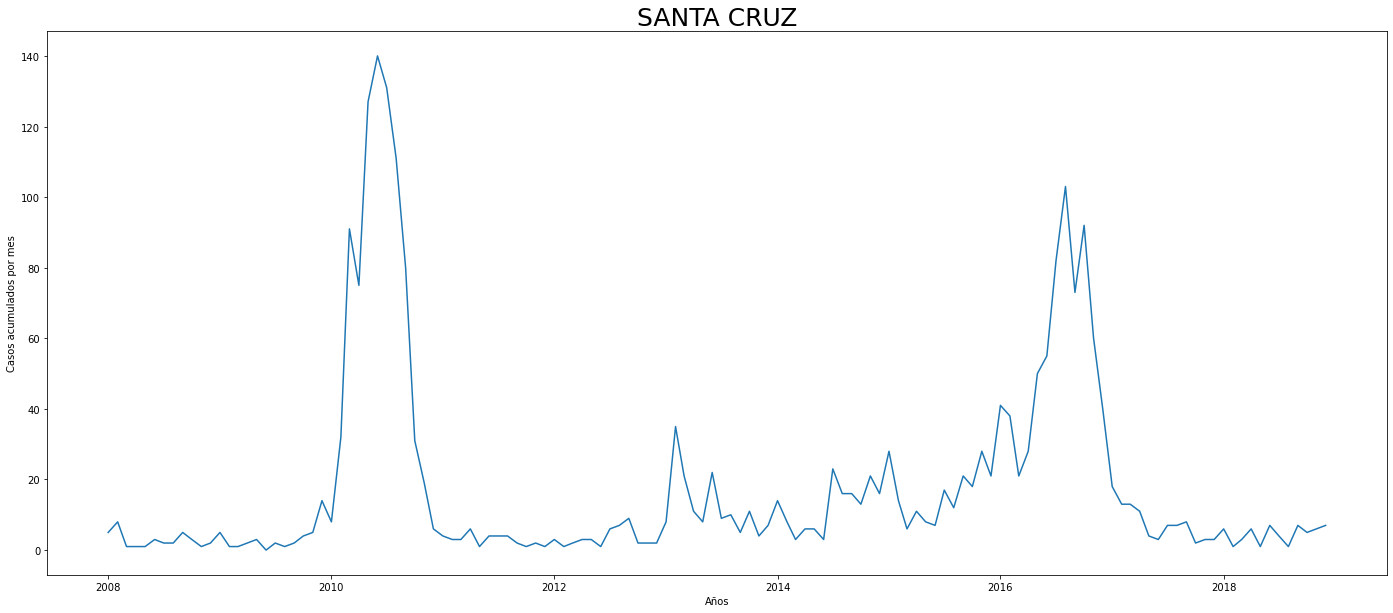
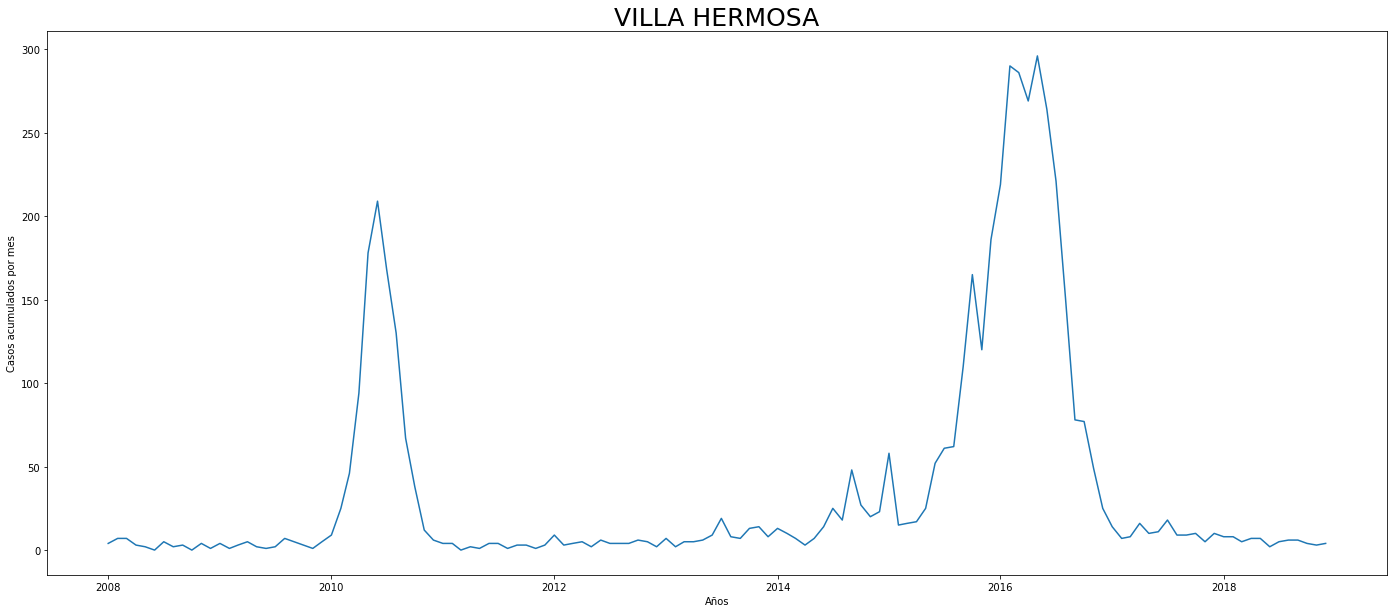
De los gráficos individuales se puede observar que:
En Altavista el pico de 2010 no es tán pronunciado, igual en San Cristobal, Santa Elena y San Antonio de Prado. Estos son corregimientos de Medellín, algo así como una ruralidad urbana. El Corregimiento de Palmitas presenta datos anómalos, por lo que se tendrá en cuenta más adelante para ser descartado.
Para el 2016 las comunas con menos casos mensuales que las demás fueron Palmitas, Santa Elena, San Cristobal, La America, El Poblado, Laureles, Guayabal y Altavista
Esto puede ser comprobado de la siguiente manera:
dengue_1016 = dengue_comuna.loc[dengue_comuna.index.year.isin([2010,2016])]
dengue_1016_resumen = dengue_1016.groupby(['comuna',dengue_1016.index.year]).sum().reset_index()
print('Sumatoria de casos por comuna para 2010')
print(dengue_1016_resumen.loc[dengue_1016_resumen.date==2010].sort_values('casos'))
print('\n\n')
print('Sumatoria de casos por comuna para 2016')
print(dengue_1016_resumen.loc[dengue_1016_resumen.date==2016].sort_values('casos'))
Sumatoria de casos por comuna para 2010
comuna date casos
36 SAN SEBASTIAN DE PALMITAS 2010 11
12 CORREGIMIENTO DE SANTA ELENA 2010 24
10 CORREGIMIENTO DE SAN CRISTOBAL 2010 102
0 ALTAVISTA 2010 165
32 SAN ANTONIO DE PRADO 2010 245
16 EL POBLADO 2010 561
18 GUAYABAL 2010 562
8 CASTILLA 2010 701
14 DOCE DE OCTUBRE 2010 707
24 LAURELES 2010 733
28 POPULAR 2010 752
22 LA CANDELARIA 2010 753
38 SANTA CRUZ 2010 851
30 ROBLEDO 2010 868
6 BUENOS AIRES 2010 953
40 VILLA HERMOSA 2010 982
20 LA AMERICA 2010 987
34 SAN JAVIER 2010 1125
26 MANRIQUE 2010 1440
2 ARANJUEZ 2010 1580
4 BELEN 2010 1864
Sumatoria de casos por comuna para 2016
comuna date casos
37 SAN SEBASTIAN DE PALMITAS 2016 4
13 CORREGIMIENTO DE SANTA ELENA 2016 113
11 CORREGIMIENTO DE SAN CRISTOBAL 2016 257
21 LA AMERICA 2016 383
17 EL POBLADO 2016 404
25 LAURELES 2016 497
19 GUAYABAL 2016 504
1 ALTAVISTA 2016 505
9 CASTILLA 2016 591
39 SANTA CRUZ 2016 683
33 SAN ANTONIO DE PRADO 2016 701
31 ROBLEDO 2016 791
3 ARANJUEZ 2016 840
29 POPULAR 2016 1043
35 SAN JAVIER 2016 1083
15 DOCE DE OCTUBRE 2016 1104
23 LA CANDELARIA 2016 1135
5 BELEN 2016 1230
27 MANRIQUE 2016 1472
7 BUENOS AIRES 2016 1851
41 VILLA HERMOSA 2016 2225
SECCIÓN A.III - EXPLORACIÓN COMO SERIE DE TIEMPO
SECCIÓN A.III.I - AJUSTE TEMPORAL
#Para continuar de la manera más sencilla agruparé a nivel mensual desde 2008 hasta 2018 la cantidad de casos.
#Dejando únicamente el índice como un objeto 'datetime' y la cantidad de casos:
dengue_mensual = dengue_tidy.resample('MS', on = 'date').sum().drop('edad_',axis=1)
print(dengue_mensual)
dengue_mensual.shape[0]/12
#Las 132 observaciones corresponden los doce meses de los 11 años que abarcan los datos
casos
date
2008-01-01 95
2008-02-01 70
2008-03-01 62
2008-04-01 46
2008-05-01 74
... ...
2018-08-01 94
2018-09-01 93
2018-10-01 106
2018-11-01 95
2018-12-01 83
[132 rows x 1 columns]
11.0
#Con las siguientes líneas de código podemos ver el comportamiento medio durante los 12 meses de todos los años
indice_mensual = dengue_mensual.index.month
dengue_pormes = dengue_mensual.groupby(indice_mensual).mean()
dengue_pormes.plot()
<AxesSubplot:xlabel='date'>
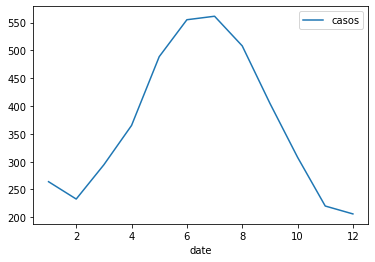
Este es el comportamiento promedio mensual. Se observa que desde abril hasta junio o julio hay aumento en los casos, luego un descenso de los contagiados. Es de recordar que esto son los casos promedios por mes por año. El comportamiento para cada año puede ser diferente, pero en promedio esta es la tendencia. Esto se puede corroborar a continuación verificando estacionalidad:
import statsmodels.api as sm
from pylab import rcParams
rcParams['figure.figsize'] = 11,9
#Lo descompondremos a nivel mensual:
decomposition = sm.tsa.seasonal_decompose(dengue_mensual ,model='additive')
fig= decomposition.plot()
plt.show()
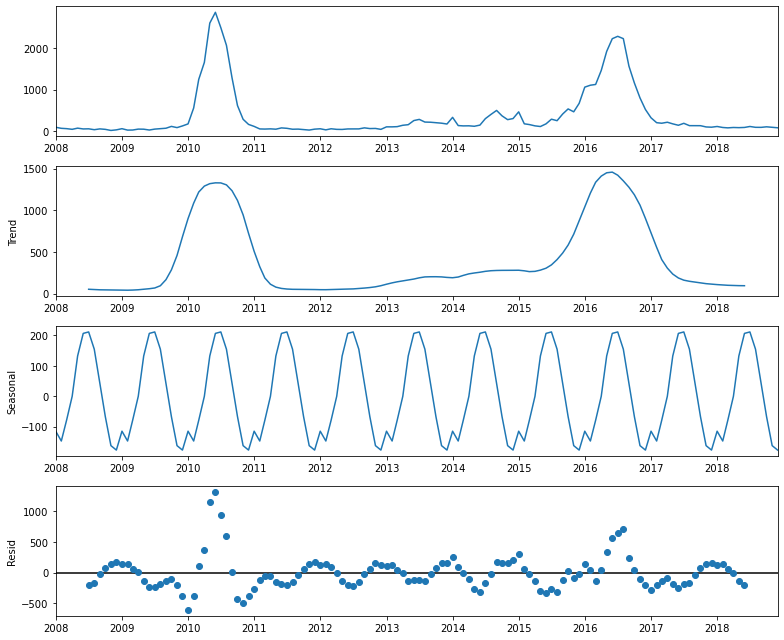
Al Observar las diferentes gráficas que proporciona el operador seasonal_decompose se reafirma la tandencia a aumentar a mediados de 2010 y 2016. Este comportamiento “outlier” sesgará fuertemente el modelo de predicción más adelante.
SECCIÓN A.III.II - AUTOCORRELACIÓN
Voy a analizar la autocorrelación de la serie con una cantidad de lags igual a la cantidad de observaciones (meses).
from statsmodels.tsa.stattools import acf
nlags = dengue_mensual.shape[0] -1
autocorr = acf(dengue_mensual.casos, nlags = nlags)
plt.figure(figsize = (16,10))
plt.scatter(x=np.arange(nlags+1), y= autocorr)
plt.title('Autocorrelación por 132 meses (11 años)')
plt.vlines(x=[range(12,132,12)],ymin=-0.3,ymax=1,colors='red')
plt.xlabel('Cantidad de lags (meses)')
Text(0.5, 0, 'Cantidad de lags (meses)')
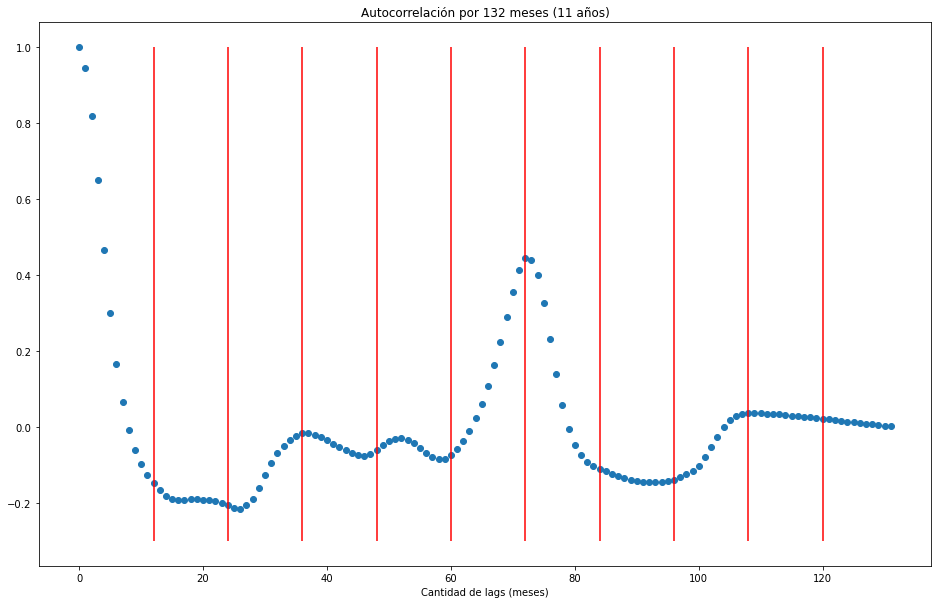
print(f"Por autocorrelación se observa que luego de casi 6 años (72 meses), hay una correlación que aumenta a un poco más de {autocorr[71]:.2f}")
Por autocorrelación se observa que luego de casi 6 años (72 meses), hay una correlación que aumenta a un poco más de 0.41
plt.figure(figsize = (25,5))
from statsmodels.graphics.tsaplots import plot_acf
plot_acf(dengue_mensual.casos, lags = nlags)
plt.show()
<Figure size 1800x360 with 0 Axes>
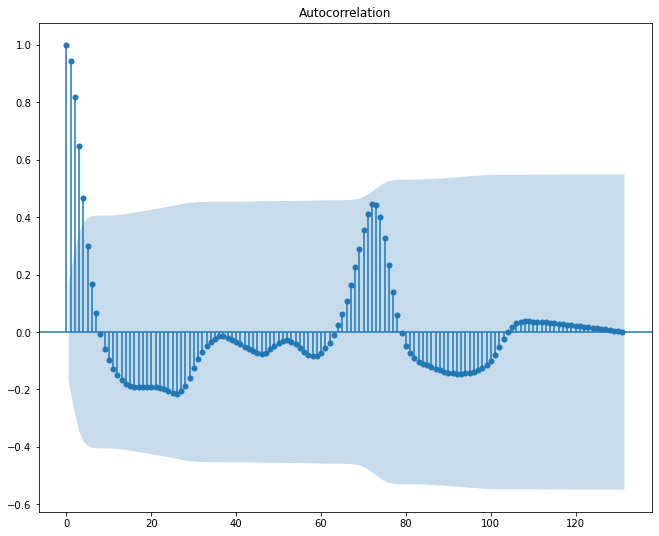
Hay mucha incertidumbre en la aucorrelación. No obastante continuaremos el ejercicio entrenando un modelo de random forest. Dejaré como entrenamiento los primeros 7 años (84 meses). Dejando los 4 años siguientes como set de testeo.
PARTE B - MODELOS DE MACHINE LEARNING
SECCIÓN B.I - TRANSFORMACIÓN DE LOS DATOS
Para realizar el modelo de Machine Learning realizaré una transformación similar a la d Rúa-Uribe et al. (2012), los autores con el fin de reducir diferencias en la serie de tiempo, aplicaron una estandarización con la media y la desviación estándar a resolución semanal.
En mi caso, lo haré a nivel mensual. Es necesario en primer lugar remuestrear a resolución diaria, esto con el fin de obtener la regularización de las fechas, así, para un solo día, aparecerán los casos acumulados y no como observaciones separadas. En segundo lugar, se obtiene la media y la desviación estándar, y por último a cada registro se le resta su media correspondiente y el resultado es dividido por la desviación estándar.
#Retirar la variable de edad que no será útil
#Resamplear a nivel de día para tener el detallado diario
#Con ese detallado diario se vuelve a resamplear a nivel mensual y se calculca la suma(total de casos), media(promedio de casos a la semana) y la desviación estándar
dengue_transform = dengue_tidy.drop('edad_',axis=1).resample('D',on = 'date').sum().reset_index().resample('MS',on ='date').agg(['sum','mean','std']).reset_index()
#El multiíndice se pasa a un índice sencillo
dengue_transform.columns = ['_'.join(col) for col in dengue_transform.columns.values]
dengue_transform.set_index('date_',inplace=True)
dengue_transform.head()
| casos_sum | casos_mean | casos_std | |
|---|---|---|---|
| date_ | |||
| 2008-01-01 | 95 | 3.166667 | 1.895245 |
| 2008-02-01 | 70 | 2.413793 | 1.239736 |
| 2008-03-01 | 62 | 2.000000 | 1.612452 |
| 2008-04-01 | 46 | 1.533333 | 1.407696 |
| 2008-05-01 | 74 | 2.387097 | 1.819843 |
#Ahora calculamos la variable a transformar que es igual a:
#casos_sum-casos_mean/casos_std
dengue_transform['casos_standard'] = (dengue_transform.casos_sum-dengue_transform.casos_mean)/dengue_transform.casos_std
dengue_standard = dengue_transform.drop(['casos_sum','casos_mean','casos_std'],axis=1)
dengue_standard.head()
| casos_standard | |
|---|---|
| date_ | |
| 2008-01-01 | 48.454595 |
| 2008-02-01 | 54.516604 |
| 2008-03-01 | 37.210420 |
| 2008-04-01 | 31.588250 |
| 2008-05-01 | 39.351147 |
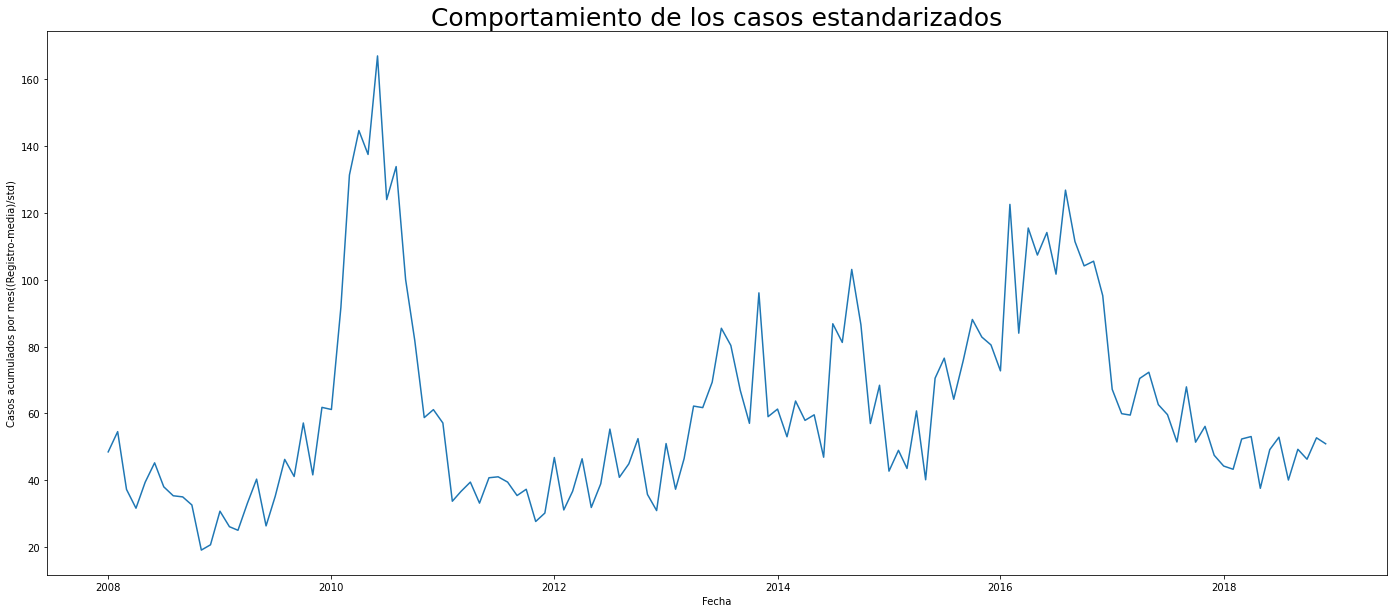
plt.figure(figsize=(24,10))
sns.lineplot(data=dengue_standard, x='date_',y='casos_standard')
plt.ylabel('Casos acumulados por mes((Registro-media)/std)')
plt.xlabel('Fecha')
plt.title('Comportamiento de los casos estandarizados',fontsize=25)
Text(0.5, 1.0, 'Comportamiento de los casos estandarizados')
SECCIÓN B.II - ENTRENAMIENTO DE MODELO RANDOM FOREST Y BÚSQUEDA DE HIPERPARÁMETROS
Para entrenar el modelo de Machine Learning utilicé como apoyo el Notebook 41 de Acámica para Ciencia de Datos del año 2020. En este ejercicio hay dos tipos de hiperparámetros (al menos así lo considero). El look_back que es la cantidad de meses hacía atrás que utilizará la ventana deslizante para calcular los próximos valores, y los hiperparámetros normales del modelo.
A continuación defino como función el train_test_split propuesto por acámica, de forma que sea reutilizable!
#Defino esta función para reutilizar el código de montar el train_test. Extraído de Acámica:
def train_test_timesplit (look_window):
N = dengue_standard.shape[0]
X = np.zeros((N - look_window - 1,look_window))
y = np.zeros(N - look_window - 1)
for i in range(X.shape[0]):
X[i,:] = np.resize(dengue_standard[i:i+look_window],look_window)
y[i] = dengue_standard.iloc[i+look_window]
N_train = int(N*0.7) - look_window
N_test = N-int(N*0.7) - look_window
N_total = N_train + N_test
length_total = N_train + N_test + look_window
X_train = X[:N_train,:]
y_train = y[:N_train]
X_test = X[N_train:N_train+N_test,:]
y_test = y[N_train:N_train+N_test]
return N, X, y, N_train, N_test, length_total, N_total, X_train, X_test, y_train, y_test
#Ejemplo para un ventana de 3 meses con un train_test de 0.6
N, X, y, N_train, N_test, length_total, N_total, X_train, X_test, y_train, y_test = train_test_timesplit(3)
El modelo a utilizar es el Random Forest, por lo que los hiperparámetros serán la profundidad, el número de estimadores y el tamaño mínimo para hacer una partición.
Para este tipo de modelo me interesa la métrica RMSE o Root Mean Squared Error, el cuál da un valor interpretable fácilmente como la raíz de la suma cuadrada de los errores dividido el número de muestras.
Además de los hiperparámetros del modelo, me planteo como hiperparámetro la cantidad de meses que toma el modelo para predecir el siguiente, el look_back, por lo que después de intentar mucho código, he decidido que esta es la mejor forma de realizarlo:
%%time
from sklearn.tree import DecisionTreeRegressor
from sklearn.metrics import r2_score, make_scorer, mean_absolute_error as mae
from sklearn.ensemble import RandomForestRegressor
from sklearn.model_selection import RandomizedSearchCV
#Defino cuántos valores hacía atrás tomaremos. Dado que estamos en resolución mensual, escojo trabjar desde 1 a 24 meses, dado que dispongo de 132 meses.
#Defino el tamaño del train_test, no son muchos, para no sezgar el modelo
look_back = [i for i in range(1,25)]
#Definimos el modelo para que no sea repetido N veces en el for
rfr = RandomForestRegressor()
rfr.get_params()
#Se usará la siguiente grilla de parámetros:
parametros_rfr = {"max_depth": [i for i in range(1,20)],
"n_estimators": [100,200,500],
"min_samples_split": range(2,12),
"random_state": [42,1111,33]}
#Recordar que para predicción se debe cambiar el score por uno de regresión.
score_rfr = make_scorer(mse)
#Para seguir de cerca las respuestas crearé estas listas
looking = []
rmse_train = []
rmse_test = []
dif_train_test = []
best_params = []
#Comienza la evaluación:
for look in look_back:
N, X, y, N_train, N_test, length_total, N_total, X_train, X_test, y_train, y_test = train_test_timesplit(look)
rfr_random = RandomizedSearchCV(estimator = rfr, param_distributions = parametros_rfr, n_iter = 100, cv = 10, verbose=2, random_state=42, n_jobs = -1,scoring=score_rfr)
# Se corre el modelo, sobre los datos de entrenamiento:
rfr_random.fit(X_train, y_train)
#Se calculan las métricas:
rfr_best_estimator = rfr_random.best_estimator_
y_train_pred_rfr = rfr_best_estimator.predict(X_train)
y_test_pred_rfr = rfr_best_estimator.predict(X_test)
rmse_train_rfr = mse(y_train, y_train_pred_rfr)
rmse_test_rfr = mse(y_test, y_test_pred_rfr)
dif_rmse = abs(rmse_train_rfr-rmse_test_rfr)
looking.append(look)
rmse_train.append(np.sqrt(rmse_train_rfr))
rmse_test.append(np.sqrt(rmse_test_rfr))
dif_train_test.append(np.sqrt(dif_rmse))
best_params.append(rfr_random.best_params_)
resultados = pd.DataFrame({'looking': looking,
'rmse_train': rmse_train,
'rmse_test' : rmse_test,
'dif_train_test' : dif_train_test,
'best_params' : best_params})
resultados
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Fitting 10 folds for each of 100 candidates, totalling 1000 fits
Wall time: 24min 54s
| looking | rmse_train | rmse_test | dif_train_test | best_params | |
|---|---|---|---|---|---|
| 0 | 1 | 17.266772 | 19.192152 | 8.378381 | {'random_state': 1111, 'n_estimators': 200, 'm... |
| 1 | 2 | 16.394206 | 17.217969 | 5.261983 | {'random_state': 42, 'n_estimators': 200, 'min... |
| 2 | 3 | 16.567620 | 17.768862 | 6.422338 | {'random_state': 42, 'n_estimators': 200, 'min... |
| 3 | 4 | 16.873551 | 19.090555 | 8.929309 | {'random_state': 33, 'n_estimators': 200, 'min... |
| 4 | 5 | 16.693530 | 19.039324 | 9.155430 | {'random_state': 1111, 'n_estimators': 200, 'm... |
| 5 | 6 | 16.795378 | 17.771302 | 5.808135 | {'random_state': 33, 'n_estimators': 100, 'min... |
| 6 | 7 | 16.970727 | 20.117997 | 10.804085 | {'random_state': 33, 'n_estimators': 100, 'min... |
| 7 | 8 | 16.809848 | 18.450114 | 7.604979 | {'random_state': 33, 'n_estimators': 100, 'min... |
| 8 | 9 | 17.213381 | 19.721648 | 9.625118 | {'random_state': 33, 'n_estimators': 100, 'min... |
| 9 | 10 | 17.129089 | 19.451853 | 9.217857 | {'random_state': 42, 'n_estimators': 200, 'min... |
| 10 | 11 | 17.141038 | 20.837039 | 11.847658 | {'random_state': 33, 'n_estimators': 100, 'min... |
| 11 | 12 | 17.048695 | 20.968324 | 12.207072 | {'random_state': 33, 'n_estimators': 200, 'min... |
| 12 | 13 | 17.021341 | 21.847726 | 13.696609 | {'random_state': 1111, 'n_estimators': 200, 'm... |
| 13 | 14 | 17.001272 | 22.164856 | 14.221026 | {'random_state': 42, 'n_estimators': 500, 'min... |
| 14 | 15 | 17.048348 | 23.448395 | 16.099100 | {'random_state': 33, 'n_estimators': 200, 'min... |
| 15 | 16 | 16.816795 | 23.252089 | 16.057865 | {'random_state': 1111, 'n_estimators': 500, 'm... |
| 16 | 17 | 17.162358 | 22.277136 | 14.202967 | {'random_state': 33, 'n_estimators': 100, 'min... |
| 17 | 18 | 17.042361 | 25.487144 | 18.951318 | {'random_state': 33, 'n_estimators': 100, 'min... |
| 18 | 19 | 16.644338 | 23.979604 | 17.262312 | {'random_state': 33, 'n_estimators': 100, 'min... |
| 19 | 20 | 16.974531 | 25.429660 | 18.934965 | {'random_state': 1111, 'n_estimators': 500, 'm... |
| 20 | 21 | 17.297444 | 26.659456 | 20.286080 | {'random_state': 42, 'n_estimators': 200, 'min... |
| 21 | 22 | 17.446037 | 27.127046 | 20.772877 | {'random_state': 42, 'n_estimators': 200, 'min... |
| 22 | 23 | 17.357261 | 26.427153 | 19.927867 | {'random_state': 1111, 'n_estimators': 200, 'm... |
| 23 | 24 | 17.510529 | 27.990057 | 21.836315 | {'random_state': 1111, 'n_estimators': 200, 'm... |
best = resultados[(resultados.rmse_train == resultados.rmse_train.min()) | (resultados.rmse_test == resultados.rmse_test.min()) | (resultados.dif_train_test == resultados.dif_train_test.min())]
best
| looking | rmse_train | rmse_test | dif_train_test | best_params | |
|---|---|---|---|---|---|
| 1 | 2 | 16.394206 | 17.217969 | 5.261983 | {'random_state': 42, 'n_estimators': 200, 'min... |
print(f"El mejor 'look_back' es : {best.looking[1]} con un RMSE en test: {best.rmse_test[1]} y los parámetros: {best.best_params[1]}")
El mejor 'look_back' es : 2 con un RMSE en test: 17.21796940840441 y los parámetros: {'random_state': 42, 'n_estimators': 200, 'min_samples_split': 5, 'max_depth': 1}
Volvemos a entrenar el modelo con los parámetros obtenidos parámetros para graficarlo:
look_back = best.looking[1]
N, X, y, N_train, N_test, length_total, N_total, X_train, X_test, y_train, y_test = train_test_timesplit(look_back)
parameters = best.best_params[1]
rf = RandomForestRegressor(**parameters).fit(X_train,y_train)
#Predecimos:
y_pred = rf.predict(X)
y_train_pred = rf.predict(X_train)
y_test_pred = rf.predict(X_test)
Y graficamos:
plt.figure(figsize=(16,8))
plt.plot(dengue_standard.index[:length_total], dengue_standard[:length_total],lw = 0.75, label = 'Serie Original')
plt.plot(dengue_standard.index[:N_train + look_back], dengue_standard[:N_train + look_back],'--', label = 'Usado para entrenar')
plt.plot(dengue_standard.index[look_back: look_back + N_train], y_train_pred,'-.',label = 'Predicho en Train')
plt.plot(dengue_standard.index[N_train+look_back: length_total], y_test_pred,'-.', lw = 4, label = 'Predicho en Test')
plt.legend()
plt.xlabel('Fecha')
plt.ylabel('Casos/mes')
plt.show()
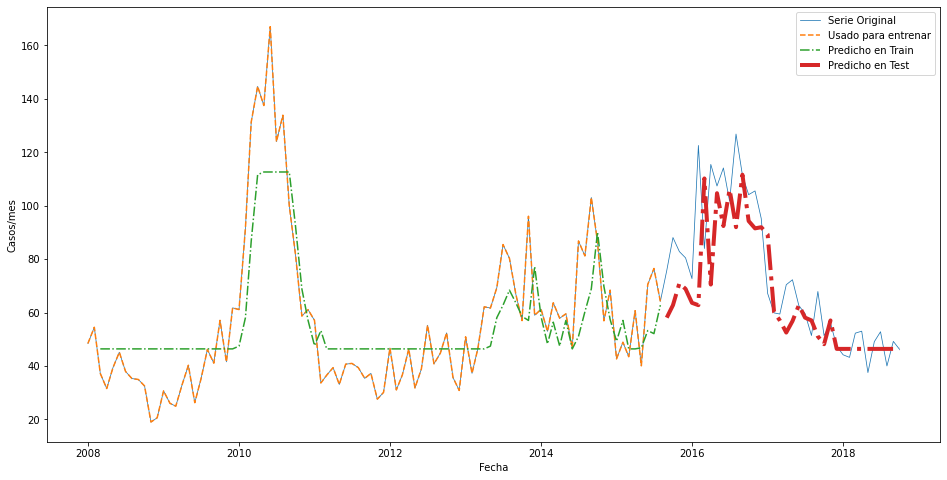
Aparentemente se ha comportado muy bien. Echemos nuevamente un vistazo a las métricas del error:
MSE_train = mse(y_train, y_train_pred)
print('Error en Train:',np.sqrt(MSE_train))
MSE_test = mse(y_test, y_test_pred)
print('Error en Test:',np.sqrt(MSE_test))
Error en Train: 16.39420630426208
Error en Test: 17.21796940840441
Benchmark: Para tener un punto de comparación con otro modelo, comparo el error con el que habríamos obtenido si predecíamos copiando el valor de la fecha anterio, que sería el modelo más básico, desplazado 1 mes en el test
y_test_pred_benchmark = y[-1 + N_train:N_train+N_test - 1]
plt.figure(figsize=(12,4))
plt.plot(dengue_standard.index[:length_total], dengue_standard[:length_total],lw = 0.75, label = 'Serie Original')
plt.plot(dengue_standard.index[N_train+look_back: length_total], y_test_pred_benchmark,'-.', lw = 2, label = 'Predicho en Test, Benchmark')
plt.xlim(dengue_standard.index[N_train+look_back], dengue_standard.index[length_total])
plt.xlabel('Fecha')
plt.ylabel('Casos/mes')
plt.legend()
plt.show()
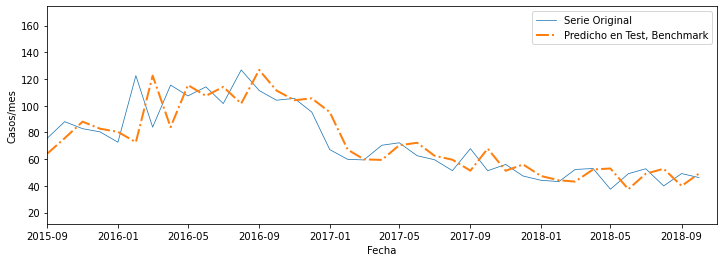
y_test_pred_benchmark = y[-1 + N_train:N_train+N_test - 1]
MSE_benchmark = mse(y_test, y[-1 + N_train:N_train+N_test - 1])
print(f"La RMSE del benchmark es: {np.sqrt(MSE_benchmark)},\n mientras la del test de random forest es {np.sqrt(MSE_test)},\n es evidente que el benchmark tiene una ventaja, pero el modelo de machine learning es mucho más poderoso.")
La RMSE del benchmark es: 15.524833122137865,
mientras la del test de random forest es 17.21796940840441,
es evidente que el benchmark tiene una ventaja, pero el modelo de machine learning es mucho más poderoso.
PARTE C - INVESTIGACIÓN
SECCIÓN C.I PREMISA
Finalmente, a modo de conclusión, espero para el proyecto 4 realizar un web scraping del “Sistema de Alerta Temprana de Medellín y el Valle de Aburrá” (SIATA) para obtener variables de precipitación y temperatura. La idea con esta información sería ajustar un modelo a variables climáticas y no a su autocorrelación en el tiempo.
Dado que el dengue es una enfermedad arboviral, y es considerada en las regiones tropicales (como es el caso de Medellín) una enfermedad epidémica con brotes endémicos (Martínez, 2008), lo más seguro es que el dengue responda a otras variables, como puede ser los fenómenos del niño y de la niña.
Existe un índice que mide el comportamiento de este fenómeno y esrecolectado por La Administración Nacional Oceánica y Atmosférica de los Estados Unidos (NOAA por sus siglas en inglés). El SOI (Southern Oscilation Index) representa una medida estandarizada de la diferencia de niveles de presión del mar entre Tahití, Polinesia Francesa y Darwin, Australia. El SOI indica una fluctuación de la presión del aire a gran escala entre el pacífico oriental y occidental. Por lo general valores negativos indican una condición cálida conocido como El Niño y valores positivos temperaturas más frías usualmente La Niña.
Para Medellín, Hales et al. (1996) encuentra que en los periodos de La Niña (valores positivos) hay un aumento de los casos, en el periodo de 1970 a 1996.
Ceballos et al. (2020) ajusta una red neuronal probablemente al mismo set de datos que estoy explorando y utiliza información del Instituto de Hidrología, Meteorología y Estudios Ambientales (IDEAM).
Se observa que hay evidencia para continuar analizando esta información. Espero poder en el próximo proyecto acercarme con más preguntas de la mano de variables que me ayuden a resolverlas. ¿Qué relación tiene las lluvias con la ocurrencia de dengue, o las temperaturas? y ¿qué tal el SOI?
SECCIÓN C.II EXPLORACIÓN CON EL SOI
En la página de la NOAA se encuentra el histórico del SOI desde 1951:
url_SOI = "https://www.ncdc.noaa.gov/teleconnections/enso/indicators/soi/data.csv"
SOI_raw = pd.read_csv(url_SOI,header=1)
print(SOI_raw.head())
print(SOI_raw.shape)
Date Value
0 195101 1.5
1 195102 0.9
2 195103 -0.1
3 195104 -0.3
4 195105 -0.7
(842, 2)
Para obtener adecuadamente la fecha debo separar el año del mes, así que me apoyo de esta función:
def insert_sting_middle(str, word):
return str[:4] + word + str[4:]
#Para tener las fechas como índice:
fechas = []
for i, element in enumerate(SOI_raw.Date):
fechas.append(insert_sting_middle(str(SOI_raw.Date[i]),'-'))
SOI_raw['fechas'] = pd.to_datetime(fechas)
SOI = SOI_raw.drop('Date',axis=1).set_index('fechas')
SOI
| Value | |
|---|---|
| fechas | |
| 1951-01-01 | 1.5 |
| 1951-02-01 | 0.9 |
| 1951-03-01 | -0.1 |
| 1951-04-01 | -0.3 |
| 1951-05-01 | -0.7 |
| ... | ... |
| 2020-10-01 | 0.5 |
| 2020-11-01 | 0.7 |
| 2020-12-01 | 1.8 |
| 2021-01-01 | 1.9 |
| 2021-02-01 | 1.5 |
842 rows × 1 columns
#Graficamos entre 2008 y 2018
SOI_medellin = SOI[SOI.index.year.isin([i for i in range(2008,2019)])]
SOI_medellin.plot()
plt.show()
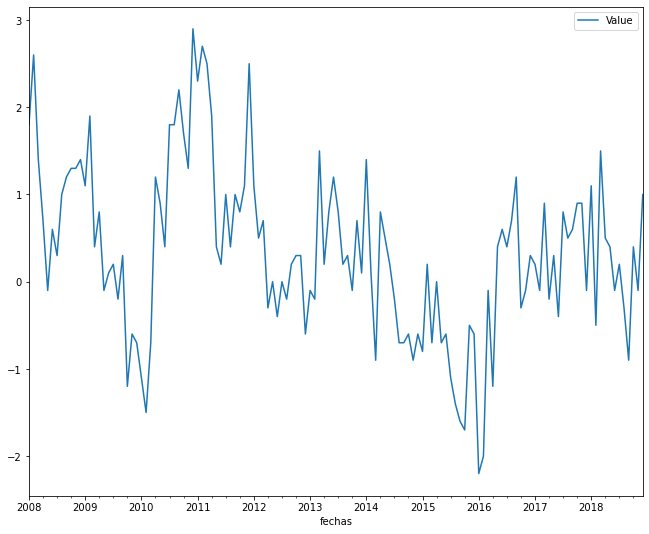
print(f"El SOI tiene {SOI_medellin.shape[0]} observaciones, y la base de dengue {dengue_standard.shape[0]}.\n Esto quiere decir que son comparables")
El SOI tiene 132 observaciones, y la base de dengue 132.
Esto quiere decir que son comparables
Miraremos brevemente la correlación entre las variables:
(f"La correlación es de {dengue_standard['casos_standard'].corr(SOI_medellin['Value'])}")
'La correlación es de -0.15800065602110072'
#Una gráfica que ayude a visualizar la información
plt.scatter(dengue_standard['casos_standard'], SOI_medellin['Value'])
plt.xlabel('Casos_estandar_dengue')
plt.ylabel('Valor del SOI')
plt.show()
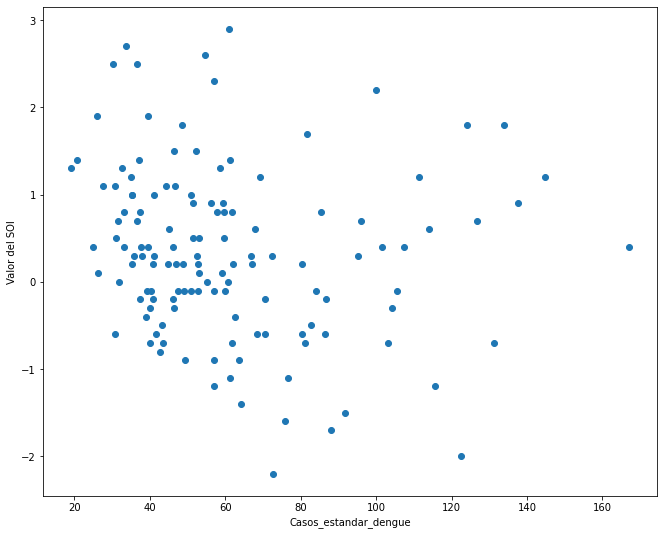
SECCIÓN C.III SOBRE LOS RESULTADOS ESPERADOS
Según el resultado de correlación, se observa un valor negativo. Esto es, que a mayor cantidad de casos de dengue menor es el valor del SOI, lo que podría ser un indicio que la mayor cantidad de casos mensuales de dengue se presentan en meses con SOI bajos (períodos EL NIÑO).
Espero en el próximo proyecto continuar con este análisis y agregar otras 4 variables: precipitación mensual (mm), temperatura máxima, mínima y media mensual (°C). Con ello podría observar alguna correlación que intente explicar la aparición del fenómeno diferente a la autocorrelación temporal del mismo.
BIBLIOGRAFÍA
- Ceballos-Arroyo, A. M., Maldonado-Perez, D., Mesa-Yepes, H., Perez, L., Ciuoderis, K., Comach, G., … & Branch-Bedoya, J. W. (2020, January). Towards a machine learning-based approach to forecasting Dengue virus outbreaks in Colombian cities: a case-study: Medellin, Antioquia. In 15th International Symposium on Medical Information Processing and Analysis (Vol. 11330, p. 1133016). International Society for Optics and Photonics.
- Hales S, Weinstein P, Woodward A. (1996). Dengue fever epidemics in the South Pacific: driven by El Niño Southern Oscillation? Lancet ;348(9042):1664–5.
- Martínez Torres, E. (2008). Dengue. Estudos avançados, 22(64), 33-52.
- Rúa Uribe, G.L., Londoño, D. A. C., Ospina, R. A. R., Correa, E. A. H., González, W. H. S., & Acosta, C. D. R. S. (2012). Influencia del evento climático El Niño sobre la dinámica de transmisión de dengue en Medellín, Antioquia, Colombia. Iatreia, 25(4), 314-322.